Kurs:Räumliche Modellbildung/Gruppe 14
Gruppenseite - (BKPR)
[Bearbeiten]Diese Seite enthält die gesammelten Materialien der Gruppe 14 für die Portfolioprüfung.
Teilnehmer-innen
[Bearbeiten]- Bentz, Judith
- Kastor, Michael Urs Lars
- Pfanger, Lara
- Renner, Hannah
Diskrete Modellierung
[Bearbeiten]Kontinuierliche Modellierung
[Bearbeiten]Tutorium 1 - Kontaktmodell
[Bearbeiten]
In Tutoriumsaufgabe I geht es um das Kontaktmodell. Das Modell soll die räumliche Ausbreitung von Covid-19 über einen selbst gewählten Zeitraum hinweg darstellen. Ausgangspunkt für das Modell ist eine Gruppe von 200 Personen. In der Mitte der Personengruppe befindet sich die erste infizierte Person, die durch einen roten Punkt in der Matrix dargestellt ist. Nachdem im Modell der Richtungswechsel eingebaut wurde, wurden mehrere verschiedene Szenarien ausprobiert, um die Auswirkungen von kleineren und größeren Ansteckungsradien, sowie schnellerer und langsamerer Bewegungen beobachten zu können. Um schrittweise Richtungswechsel der Personen in die Matrix zu implementieren, wurde eine for-Schleife in das Jupyter-Notebook eingefügt. Durch die for-schleife wird die Bewegungsrichtung der Personen in jedem Zeitschritt zufällig geändert (siehe GIF).Der unten dargestellte Programmcode entstand durch Bearbeitung des von Frau Prof. Dr. Hundertmark zur Verfügung gestellten Codes.
Modellierung der Bevölkerung
[Bearbeiten]%Anzahl von Personen in x- und y-Richtung Nx=10; Ny=20; Zeitschritte=2; %pro Zeiteinheit T radiusInf=0.80; %Infektionsradius T=1; %Zeitspanne, in der sich Personen nur in eine Richtung bewegen g=0.50; %Geschwindigkeit der Fußgänger W=8 %Anzahl der Richtungswechsel %------------------------------------------------------------------------------------------------- x=[1:Nx]; y=[1:Ny]; dt=1/Zeitschritte;
[x,y]=meshgrid (x,y)
Erzeugen des ersten Infizierten
[Bearbeiten]%Erste Infizierte Person xInf=x(1, Nx/2); yInf=y(Ny/2); IndInf2=Nx/2; %Spalte IndInf1=Ny/2; %Zeile IndInf1neu=IndInf1; IndInf2neu=IndInf2;
close all;
%Darstellung des Anfangszustandes figure (1) hold on; plot(x,y, '*b') ; plot(xInf,yInf, 'sr') ; axis([-5 15 -5 25]);
hold off;
Erzeugen zufälliger Richtungsänderungen
[Bearbeiten]Anzahl=0; %Zur zeitlichen Darstellung der Infiziertenzahl
neuX=x; neuY=y; z=1; %Schleife zur wiederholten Richtungsänderung
for k=1:W
neuPosX=g.*rand(Ny,Nx)-0.5*g; neuPosY=g.*rand(Ny,Nx)-0.5*g;
%------------------------------------------------------------------------------------------------- %Zeitschleife für kurzzeitige Bewegung mit konstanter Geschwindigkeit for i=1:T*Zeitschritte
%Neue Position immer relativ zu aktueller Position neuX=neuX.+neuPosX.*dt; neuY=neuY.+neuPosY.*dt; z=z+1;%Zählparameter für die Erstellung der Bilder
%Darstellung der Personen zum Zeitpunkt (z-1)*dt figure (z) title (['t=' num2str((z-1)*dt)]) axis([-5 15 -5 25]); hold on plot(neuX,neuY, '*b') ; quiver (neuX,neuY, neuPosX, neuPosY); %Geschwindigkeitspfeile
Erzeugen neuer Infizierter durch Ansteckung
[Bearbeiten]%Neue Infizierungen: %In den Vektoren IndInf1, IndInf2 sind die i- und j-Indizes der (x,y)-Koordinaten von den bereits Infizierten gespeichert. %-------------------------------------------------------------------------------------------------
zahler=1; for k1=1:length(IndInf1) for j=1:Nx for l=1:Ny %Die aktuellen Positionen der Fußgänger-Punkte werden mit den Positionen der Infizierten abgeglichen (Abstand wird berechnet abstand=norm ( [neuX(l,j)-neuX(IndInf1(k1),IndInf2(k1)), neuY(l,j)-neuY(IndInf1(k1),IndInf2(k1))]); if abstand<radiusInf %wenn der Abstand zum Infizierten unter dem Ansteckungradius liegt, wird der l,j-te Punkt infiziert (mit rotem Quadrat gekennzeichnet) abstand; plot(neuX(l,j),neuY(l,j), 'sr') ; %-------------------------------------------------------------------------------------- k2=1; %Der Vektor der Infizierten wird untersucht, falls die l,j-Indizes des aktuellen Punkten nicht unter den %infizierten Indizes auftauchen, werden die l,j-Indizes des aktuell Infizierten zu den neuen Vektoren der Infizierten-Indizes %IndInf1neu, IndInf2neu zugefügt. %(Grund: Speicheroptimierung) while k2 <=length(IndInf1)&&(l!=IndInf1neu(k2) || j!=IndInf2neu(k2)) k2=k2+1; endwhile
if k2 >length(IndInf1) IndInf1neu=[IndInf1neu l]; IndInf2neu=[IndInf2neu j]; endif endif IndInf1=IndInf1neu; IndInf2=IndInf2neu; endfor endfor
endfor
%Speichern der Bilder test=["Fig1_", num2str(z),".jpg"] saveas(z, test)
hold off
lengt=length(IndInf1) Anzahl= [Anzahl lengt];
endfor endfor %Zeitschleife abgeschlossen %------------------------------------------------------------------------------------------------- Infiz1=[IndInf1; IndInf2] %Ausgabe Infizierten-Indizes hold off;
Modellierung des Kontaktmodells mit höherer Geschwindigkeit
[Bearbeiten]
Nachfolgend wurde die Übertragung der Infektion für eine höhere Bewegungsgeschwindigkeit modelliert. Die Infizierten behalten den Richtungswechsel in jedem Zeitschritt bei. Zur Erhöhung der Bewegungsgeschwindigkeit wurde der Parameter "Param" eingeführt, sodass die Geschwindigkeit ohne große Umstände beliebig variiert werden kann. In diesem Fall wurde die Geschwindigkeit vervierfacht. Die Veränderung der Infektionsübertragung bei einer vierfachen Bewegungsgeschwindigkeit ist im nebenstehenden GIF zu sehen.
Param= 4.0;
%Aktualisierung, erste Infizierte in Infiziertenindexen xInf=x(1, Nx/2); yInf=y(Ny/2); IndInf2=Nx/2; IndInf1=Ny/2; IndInf1neu=IndInf1; IndInf2neu=IndInf2;
%---------------------------------- %Zeitschleife II %--------------------------- Anzahl2=0; % Zur zeitlichen Darstellung der Infiziertenzahl
neuX=x; neuY=y; z=1;
for k=1:W
neuPosX=g.*rand(Ny,Nx)-0.5*g; neuPosY=g.*rand(Ny,Nx)-0.5*g;
%Zeitschleife für 2 Zeitschritte pro eine Zeiteinheit T: %--------------------------- for i=1:T*Zeitschritte
%neue Position immer relativ zu aktueller Position neuX=neuX.+Param*neuPosX.*dt; neuY=neuY.+Param*neuPosY.*dt; z=z+1;
figure (z) title (['t=' num2str((z-1)*dt)]) axis([-5 15 -5 25]); hold on plot(neuX,neuY, '*b') ; quiver (neuX,neuY, neuPosX, neuPosY);
%neue Infizierungen: %In Vektoren IndInf1, IndInf2 sind die i- und j-Indexen der (x,y)-Koordinaten der bereits Infizierten gespeichert. %---------------------------
zahler=1; for k1=1:length(IndInf1) for j=1:Nx for l=1:Ny %die aktuellen Positionen der Füßgänger-Punkte werden mit den Positionen der Infizierten abgeglichen (Abstand wird berechnet) abstand=norm ( [neuX(l,j)-neuX(IndInf1(k1),IndInf2(k1)), neuY(l,j)-neuY(IndInf1(k1),IndInf2(k1))]); if abstand<radiusInf %wenn der Abstand zum Infizierten unter dem Ansteckunsradius liegt, wird der l,j-te Punkt infiziert (mit rotem Quadrat gekenzeichnet) abstand; plot(neuX(l,j),neuY(l,j), 'sr') ; %--------------------------- k2=1; % der Vektor der Infizierten wird untersucht, falls die l,j-Indexen des aktuellen Punkten nicht unter den %infizierten Indexen auftauchen, werden die l,j-Indexen des aktuell Infizierten zu den neuen Vektoren der Infizierten-Indexen % IndInf1neu, IndInf2neu zugefügt. %(Grund: Speicheroptimierung) while k2 <=length(IndInf1)&&(l!=IndInf1neu(k2) || j!=IndInf2neu(k2)) k2=k2+1; endwhile
if k2 >length(IndInf1) IndInf1neu=[IndInf1neu l]; IndInf2neu=[IndInf2neu j]; endif endif IndInf1=IndInf1neu; IndInf2=IndInf2neu; endfor endfor
endfor
test=["Fig2_", num2str(z),".jpg"] saveas(z, test)
hold off
Anzahl2= [Anzahl2 length(IndInf1)];
endfor endfor %Zeitschleife abgeschlossen %---------------------------
hold off;
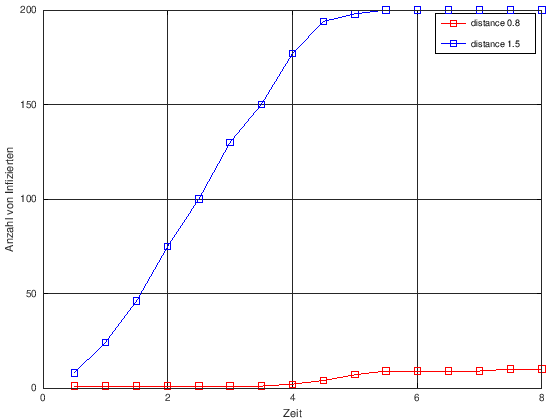
Das Diagramm zeigt zudem den Vergleich zwischen der Infektionsverbreitung bei ursprünglicher und vervierfachter Bewegungsgeschwindigkeit. Zu sehen ist, dass die langsamere Ausbreitung ("slower" - roter Graph) und die schnellere Ausbreitung ("faster" - blauer Graph) bis zur vierten Zeiteinheit ähnlich verlaufen. Ab der vierten Zeiteinheit unterscheiden sich die Infektionsausbreitungen der jeweiligen Bewegungsgeschwindigkeit deutlich voneinander. In der achten und somit letzten modellierten Zeiteinheit sind bei langsamerer Geschwindigkeit 10 Personen infiziert, während bei der vierfachen Geschwindigkeit bereits 74 Personen infiziert wurden.
Modellierung des Kontaktmodells mit verändertem Abstand
[Bearbeiten]
Die Infektionsübertragung mit verändertem Abstand wurde ebenfalls modelliert. Auch hier wechselt sich die Richtung der Personen in jedem Zeitschritt. Zur Veränderung des Abstands wurde im Programmcode der dafür vorgesehene Parameter radiusInf auf 1,5 m erhöht. Die damit einhergehende Änderung der Infektionsausbreitung ist im GIF zu erkennen. Hier erkennt man eine deutlich schnellere Ausbreitung der Infektion als im GIF der ursprünglichen Modellierung, bei der eine Person ab einem Radius von 0,8 m infiziert werden kann.
Das Diagramm visualisiert den Vergleich zwischen variierten Radien, ab denen es zu einer Übertragung der Infektion kommt. Zu erkennen ist, dass der Verlauf der Ausbreitung schon ab dem ersten Zeitschritt unterschiedlich ist. Der blaue Graph gibt dabei die Infektionsübertragung ab einem Radius von 1,5m an, der rote Graph eine Übertragung ab einem Radius von 0,8 m. Bei einem größeren Radius werden mehr Personen in deutlich schnellerer Zeit angesteckt. Bereits vor der sechsten Zeiteinheit sind alle 200 Personen mit Covid-19 infiziert. Während bei einer Übertragung der Infektion ab einem Radius von 0,8 m in der achten und somit letzten modellierten Zeiteinheit lediglich 10 Personen infiziert sind.
Tutorium 2 - Fundamentallösungen der Diffusionsgleichung
[Bearbeiten]In Tutoriumsaufgabe II geht es um die Diffusion. Da sich die Brown'sche Bewegung von Teilchen und die zufälligen Bewegungen von Menschen ähneln, werden in dieser Aufgabe zunächst die Erkenntnisse aus dem Bereich der Diffusion angewendet. Konkret bedeutet dies, dass hier die Fundamentallösungen der Laplace- und Diffusionsgleichung dargestellt werden. Diese werden anschließend genutzt, um die Poissongleichung für eine selbst ausgewählte Inhomogenität und die zeitabhängige Diffusionsgleichung für eine selbstgewählte Anfangsfunktion zu lösen. Der unten dargestellte Programmcode entstand durch Bearbeitung des von Frau Prof. Dr. Hundertmark zur Verfügung gestellten Codes.
Definition des Gitters
[Bearbeiten]Es wird ein rechteckiges Gebiet und ein entsprechendes Gitter mit der Schrittweite 0,3 definiert.
step=0.03; X=[0:step:2]; Y=[0:step:2]; [x,y]=meshgrid(X,Y);
Fundamentallösungen der Laplace-Gleichung und Diffusionsgleichung
[Bearbeiten]Daraufhin sollten in diesem Gebiet die Fundamentallösungen der Laplace Gleichung sowie der Diffusionsgleichung graphisch dargestellt werden.
Die Laplace-Gleichung beschreibt eine stationäre Diffusion. Das bedeutet, dass die Diffusion mit einer konstanten Geschwindigkeit und unabhängig der Zeit sattfindet. Die rechte Seite der Gleichung ist gleich Null, demzufolge existiert kein Infektionsherd. Die Laplace-Gleichung besitzt folgende Formeln für die Fundamentallösung auf : ,
während das Volumen der Einheitskugel angibt und die Vektornorm in ist.

Zur graphischen Ausgabe der Fundamentallösung in wurde diese definiert und dann im entsprechenden Gitter geplottet.
phi=-log(sqrt(x.^2+y.^2))/(2*pi);
figure 4
surfc (x,y,phi)
title ('phi Aufgabe 1')
Die Fundamentallösung der Laplace-Gleichung ist für die Modellierung von Covid-19 sehr hilfreich. Wie der Abbildung auf der rechten Seite entnommen werden kann, gibt es einen starken Anstieg am Rand der graphischen Darstellung. Hierbei handelt es sich im Kontext des Infektionsgeschehens um einen sogenannten 'Hotspot' in dem es besonders viele Infizierte gibt. Umso weiter man sich von dem Punkt entfernt, desto mehr flacht die Abbildung ab und wir finden weniger Infizierte vor.
Die Diffusionsgleichung , mit und konstantem Diffusionskoeffizient beschreibt eine homogene, instationäre, zeitabhängige Diffusion. Die Diffusionsgeschwindigkeit ist demnach zeitabhängig. Die Diffusionsgleichung besitzt folgende Formel für die Fundamentallösung in :
während der euklidischen Norm von im Quadrat entspricht.

Zur graphischen Ausgabe der Fundamentallösung in wurde diese definiert und dann im entsprechenden Gitter geplottet.
psi=1/(4*pi*1*0.5)^(2/2)*exp(-(x.^2+y.^2)/(4*1*0.5)); %psi zum Zeitpunkt t=0.5 mit Diffusionskonstante 1
figure 5
surfc (x,y,psi)
title ('psi Aufgabe 1')
Definition der Quellfunktion
[Bearbeiten]
Dann wurde eine Quellfunktion definiert, die in unserem Skript einerseits als Funktion der rechten Seite der Poissongleichung und andererseits als Anfangsbedingung der Diffusionsgleichung dient.
function wert=Quellfunktion(x,y) if sqrt((x.-1.5).^2+(y.-1).^2)<=0.15 wert=1; elseif sqrt((x.-1.8).^2+(y.-1).^2)<=0.1 wert=1; elseif sqrt((x.-1.5-0.707*0.3).^2+(y.-1-0.707*0.3).^2)<=0.1 wert=1; elseif sqrt((x.-1.5-0.707*0.3).^2+(y.-1+0.707*0.3).^2)<=0.1 wert=1; elseif sqrt((x.-1.5).^2+(y.-0.7).^2)<=0.1 wert=1; elseif sqrt((x.-1.5).^2+(y.-1.3).^2)<=0.1 wert=1; elseif sqrt((x.-1.5+0.707*0.3).^2+(y.-1-0.707*0.3).^2)<=0.1 wert=1; elseif sqrt((x.-1.5+0.707*0.3).^2+(y.-1+0.707*0.3).^2)<=0.1 wert=1; else wert=0; endif if 0.9<y && y<1.1 && 0.2<x && x<1.2 wert=1; endif
endfunction
Die gewählte Funktion stellt keinen konkreten Anwendungsbezug her. Sie wurde aufgrund ihrer graphischen Darstellung gewählt. Wie der Abbildung entnommen werden kann, handelt es sich um eine Blume. Diese Blume würde dann mithilfe der Diffusion 'verwischen'.
Implementierung Poissongleichung
[Bearbeiten]Die Poissongleichung auf ist die inhomogene Gleichung der Laplace-Gleichung. Die Funktion ist zweimal stetig differenzierbar und hat einen kompakten Träger. Die Lösung der Poissonformel wird durch Faltung der Fundamentallösung und Funktion der rechten Seite erhalten:

Zur Implementierung der Poissongleichung mit der Quellfunktion als rechte Seite wurden auf dem definierten Gitter die Funktionswerte der Quellfunktion in jedem Gitterpunkt gespeichert. Dann wurde die Poissonformel implementiert und als Grafik geplottet.
step=0.03; X=[0:step:2]; Y=[0:step:2]; [x,y]=meshgrid(X,Y);
for i=1:(length(X)) for j=1:(length(Y)) f(j,i)=1*Quellfunktion(x(j,i),y(j,i)); endfor endfor
for i=1:(length(X)) for j=1:(length(Y)) xstar=x(j,i)*ones(length(Y),length(X)); ystar=y(j,i)*ones(length(Y),length(X)); % Phi mit verschobenem Argument: phi=-log(sqrt((xstar.-x).^2+(ystar.-y).^2))/(2*pi); phi(j,i)=0;
%numerische Integration I(j,i) = trapz(Y,trapz(X,phi.*f,2)); endfor endfor
phi=-log(sqrt(x.^2+y.^2))/(2*pi);
figure 1 meshc(x,y,I) grid on title ('Losung von Poissongleichung') xlabel('x') xlabel ('y')
Implementierung zeitabhängige Diffusionsgleichung
[Bearbeiten]Ein Anfangswertproblem erhält man, indem man die Diffusionsgleichung mit einer Anfangsbedingung ergänzt:
Diese Anfangsbedingung beschreibt im Anfangspunkt die Konzentrationsdichte bzw. die Dichte der Infizierten. Für das homogene Anfangswertproblem für ist die Lösung durch die Faltung der Fundamentallösung mit der Anfangsfunktion gegeben:

Zur Implementierung der Diffusionsgleichung mit der Quellfunktion als Anfangsbedingung wurden wie zuvor auf dem definierten Gitter die Funktionswerte der Quellfunktion in jedem Gitterpunkt gespeichert. Dann wurde die Diffusionsgleichung implementiert und als Grafik geplottet. Das GIF zeigt den Verlauf der Diffusionsgleichung im festgelegten Zeitschritt (0,002).
for k=1:15
for i=1:(length(X)) for j=1:(length(Y)) xstar=x(j,i)*ones(length(Y),length(X)); ystar=y(j,i)*ones(length(Y),length(X)); %psi mit verschobenem Argument: Zeitschritt=0.002; psi=1/(4*pi*1*Zeitschritt*k)^(2/2)*exp(-((xstar.-x).^2+(ystar-y).^2)/(4*1*Zeitschritt*k));
%numerische Integration J(j,i) = trapz(Y,trapz(X,psi.*f,2)); endfor endfor
figure (5+k) surfc (x,y,J) title (['Aufgabe 2b: Loesung der Diffussionsgleichung bei t=' num2str(k*Zeitschritt)]) axis([0 2 0 2 0 0.9]);
%Speichern der Bilder: test=["Fig_", num2str(5+k),".jpg"] saveas(5+k, test)
endfor
Tutorium 3 - SIR-Modell
[Bearbeiten]Das Ziel der Tutoriumsaufgabe III ist, das Gesamt-Infektionsgeschehen in Deutschland mithilfe des SIR-Modells nachzubilden. Der unten dargestellte Programmcode entstand durch Bearbeitung des von Frau Prof. Dr. Hundertmark zur Verfügung gestellten Codes. Hierzu werden zunächst die Daten des RKI bezüglich der täglichen Infizierten-, Genesenen-und Todeszahlen im Zeitraum vom 25.02.2020 bis 24.05.2021 in einer großen Funktion "coronaData" implementiert.
function y= coronaData ()
infects = [16.0 18.0 21.0 26.0 53.0 66.0 117.0 150.0 188.0 240.0 400.0 639.0 795.0 902.0 1139.0 1296.0 1567.0 2369.0 3062.0 3795.0 4838.0 6012.0 7156.0 8198.0 10999.0 13957.0 16662.0 18610.0 22672.0 27436.0 31554.0 36508.0 42288.0 48582.0 52547.0 57298.0 61913.0 67366.0 73522.0 79696.0 85778.0 91714.0 95391.0 99225.0 103228.0 108202.0 113525.0 117658.0 120479.0 123016.0 125098.0 127584.0 130450.0 133830.0 137439.0 139897.0 141672.0 143457.0 145694.0 148046.0 150383.0 152438.0 154175.0 155193.0 156337.0 157641.0 159119.0 160758.0 161703.0 162496.0 163175.0 163860.0 164807.0 166091.0 167300.0 168531.0 169218.0 169575.0 170508.0 171306.0 172239.0 173152.0 173772.0 174355.0 174697.0 175210.0 176007.0 176752.0 177212.0 177850.0 178281.0 178570.0 179002.0 179364.0 179717.0 180458.0 181196.0 181482.0 181815.0 182028.0 182370.0 182764.0 183271.0 183678.0 183979.0 184193.0 184543.0 184861.0 185416.0 185674.0 186022.0 186269.0 186461.0 186839.0 187184.0 187764.0 188534.0 189135.0 189822.0 190359.0 190862.0 191449.0 192079.0 192556.0 193243.0 193499.0 193761.0 194259.0 194725.0 195228.0 195674.0 196096.0 196335.0 196554.0 196944.0 197341.0 197783.0 198178.0 198556.0 198804.0 198963.0 199375.0 199726.0 200260.0 200843.0 201372.0 201574.0 201823.0 202345.0 202799.0 203368.0 204183.0 204964.0 205269.0 205609.0 206242.0 206926.0 207828.0 208698.0 209653.0 209893.0 210402.0 211281.0 212022.0 213067.0 214214.0 215336.0 215891.0 216327.0 217293.0 218519.0 219964.0 221413.0 222828.0 223453.0 224014.0 225404.0 226914.0 228621.0 230048.0 232082.0 232864.0 233575.0 234853.0 236429.0 237936.0 239507.0 240986.0 241771.0 242381.0 243599.0 244855.0 246166.0 247619.0 248997.0 249985.0 250799.0 252298.0 253474.0 255366.0 256850.0 258480.0 259428.0 260355.0 261762.0 263663.0 265857.0 267773.0 270070.0 271415.0 272337.0 274158.0 275927.0 278070.0 280223.0 282730.0 284140.0 285332.0 287421.0 289219.0 291722.0 294395.0 296958.0 299237.0 300619.0 303258.0 306086.0 310144.0 314660.0 319381.0 322864.0 325331.0 329453.0 334585.0 341223.0 348557.0 356387.0 361974.0 366299.0 373167.0 380762.0 392049.0 403291.0 418005.0 429181.0 437866.0 449275.0 464239.0 481013.0 499694.0 518753.0 532930.0 545027.0 560379.0 577593.0 597583.0 619089.0 642488.0 658505.0 671868.0 687200.0 705687.0 727553.0 751095.0 773556.0 790503.0 801327.0 815746.0 833307.0 855916.0 879564.0 902528.0 918269.0 929133.0 942687.0 961320.0 983588.0 1006394.0 1028089.0 1042700.0 1053869.0 1067473.0 1084743.0 1106789.0 1130238.0 1153550.0 1171323.0 1183655.0 1197709.0 1218524.0 1242203.0 1272078.0 1300516.0 1320716.0 1337078.0 1351510.0 1379238.0 1406161.0 1439938.0 1471238.0 1494009.0 1510652.0 1530180.0 1554920.0 1587115.0 1612648.0 1627103.0 1640858.0 1651834.0 1664726.0 1687185.0 1719737.0 1742661.0 1755351.0 1765666.0 1775513.0 1787410.0 1808647.0 1835038.0 1866887.0 1891581.0 1908527.0 1921024.0 1933826.0 1953426.0 1978590.0 2000958.0 2019636.0 2033518.0 2040659.0 2052028.0 2068002.0 2088400.0 2106262.0 2122679.0 2134936.0 2141665.0 2148077.0 2161275.0 2178828.0 2192850.0 2205171.0 2216363.0 2221971.0 2228085.0 2237790.0 2252001.0 2264909.0 2275394.0 2284010.0 2288545.0 2291924.0 2299996.0 2310233.0 2320093.0 2328447.0 2334561.0 2338987.0 2342843.0 2350399.0 2360606.0 2369719.0 2378883.0 2386559.0 2390928.0 2394811.0 2402818.0 2414687.0 2424684.0 2434446.0 2442336.0 2447068.0 2451011.0 2460030.0 2471942.0 2482522.0 2492079.0 2500182.0 2505193.0 2509445.0 2518591.0 2532947.0 2545781.0 2558455.0 2569245.0 2575849.0 2581329.0 2594764.0 2612268.0 2629750.0 2645783.0 2659516.0 2667225.0 2674710.0 2690523.0 2713180.0 2734753.0 2755225.0 2772401.0 2782273.0 2791822.0 2808873.0 2833173.0 2855061.0 2873190.0 2885386.0 2893883.0 2900768.0 2910445.0 2930852.0 2956316.0 2980413.0 2998268.0 3011513.0 3022323.0 3044016.0 3073442.0 3099273.0 3123077.0 3142262.0 3153699.0 3163308.0 3188192.0 3217710.0 3245253.0 3268645.0 3287418.0 3299325.0 3310301.0 3332532.0 3357268.0 3381597.0 3400532.0 3416822.0 3425982.0 3433516.0 3451550.0 3473503.0 3491988.0 3507673.0 3520329.0 3527251.0 3533376.0 3548285.0 3565704.0 3577040.0 3584934.0 3593434.0 3598846.0 3603055.0 3614095.0 3626393.0 3635162.0 3642244.0 3648958.0 3651640.0];
deads = [0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 12.0 20.0 31.0 46.0 55.0 86.0 114.0 149.0 198.0 253.0 325.0 389.0 455.0 583.0 732.0 872.0 1017.0 1158.0 1342.0 1434.0 1607.0 1861.0 2107.0 2373.0 2544.0 2673.0 2799.0 2969.0 3254.0 3569.0 3868.0 4110.0 4294.0 4404.0 4598.0 4879.0 5094.0 5321.0 5500.0 5640.0 5750.0 5913.0 6115.0 6288.0 6481.0 6575.0 6649.0 6692.0 6831.0 6996.0 7119.0 7266.0 7369.0 7395.0 7417.0 7533.0 7634.0 7723.0 7824.0 7881.0 7914.0 7935.0 8007.0 8090.0 8147.0 8174.0 8216.0 8247.0 8257.0 8302.0 8349.0 8411.0 8450.0 8489.0 8500.0 8511.0 8522.0 8551.0 8581.0 8613.0 8646.0 8668.0 8674.0 8711.0 8729.0 8755.0 8763.0 8781.0 8787.0 8791.0 8800.0 8830.0 8856.0 8872.0 8883.0 8882.0 8885.0 8895.0 8914.0 8927.0 8948.0 8954.0 8957.0 8961.0 8973.0 8985.0 8994.0 9003.0 9010.0 9012.0 9016.0 9024.0 9036.0 9048.0 9054.0 9060.0 9063.0 9064.0 9068.0 9071.0 9078.0 9082.0 9083.0 9084.0 9086.0 9090.0 9095.0 9101.0 9111.0 9118.0 9118.0 9118.0 9122.0 9128.0 9134.0 9141.0 9148.0 9141.0 9148.0 9156.0 9168.0 9175.0 9183.0 9195.0 9196.0 9197.0 9201.0 9207.0 9211.0 9225.0 9231.0 9231.0 9232.0 9236.0 9243.0 9253.0 9260.0 9267.0 9269.0 9272.0 9277.0 9280.0 9285.0 9288.0 9289.0 9295.0 9298.0 9302.0 9313.0 9321.0 9322.0 9324.0 9325.0 9325.0 9329.0 9338.0 9341.0 9342.0 9347.0 9349.0 9350.0 9362.0 9368.0 9371.0 9378.0 9384.0 9386.0 9386.0 9396.0 9409.0 9428.0 9443.0 9452.0 9457.0 9460.0 9471.0 9488.0 9500.0 9508.0 9527.0 9529.0 9534.0 9546.0 9562.0 9578.0 9589.0 9604.0 9615.0 9621.0 9634.0 96770 9710.0 9734.0 9767.0 9777.0 9789.0 9836.0 9875.0 9905.0 9954.0 10003.0 10032.0 10056.0 10098.0 10183.0 10272.0 10349.0 10452.0 10481.0 10530.0 10661.0 10812.0 10930.0 11096.0 11226.0 11289.0 11352.0 11506.0 11767.0 11982.0 12200.0 12378.0 12485.0 12547.0 12814.0 13119.0 13370.0 13630.0 13884.0 14022.0 14112.0 14361.0 14771.0 15160.0 15586.0 15965.0 16123.0 16248.0 16636.0 17123.0 17602.0 18034.0 18517.0 18772.0 18919.0 19342.0 19932.0 20372.0 20970.0 21466.0 21787.0 21975.0 22475.0 23427.0 24125.0 24938.0 25640.0 26049.0 26275.0 27006.0 27968.0 28770.0 29182.0 29422.0 29778.0 30126.0 30978.0 32107.0 33071.0 33624.0 33960.0 34272.0 34574.0 35518.0 36537.0 37607.0 38795.0 39878.0 40343.0 40686.0 41577.0 42637.0 43881.0 44994.0 45974.0 46419.0 46633.0 47622.0 48770.0 49783.0 50642.0 51521.0 51870.0 52087.0 52990.0 53972.0 54913.0 55752.0 56546.0 56945.0 57120.0 57981.0 58956.0 59742.0 60597.0 61286.0 61517.0 61675.0 62156.0 62969.0 63635.0 64191.0 64742.0 64960.0 65076.0 65604.0 66164.0 66698.0 67206.0 67696.0 67841.0 67903.0 68318.0 68740.0 69125.0 69519.0 69888.0 70045.0 70105.0 70463.0 70881.0 71240.0 71504.0 71804.0 71900.0 71934.0 72189.0 72489.0 72810.0 73062.0 73301.0 73371.0 73418.0 73656.0 73905.0 74132.0 74358.0 74565.0 74664.0 74714.0 74964.0 75212.0 75440.0 75623.0 75780.0 75870.0 75913.0 76093.0 76342.0 76543.0 76775.0 76895.0 76963.0 77013.0 77103.0 77401.0 77707.0 78003.0 78249.0 78353.0 78452.0 78746.0 79088.0 79381.0 79628.0 79847.0 79914.0 80006.0 80303.0 80634.0 80893.0 81158.0 81444.0 81564.0 81624.0 81968.0 82280.0 82544.0 82850.0 83082.0 83192.0 83276.0 83591.0 83876.0 84126.0 84410.0 84648.0 84775.0 84829.0 85112.0 85380.0 85658.0 85848.0 86025.0 86096.0 86160.0 86381.0 86665.0 86902.0 87128.0 87298.0 87380.0 87423.0];
recovered = [0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 0.0 13500.0 16100.0 18700.0 21400.0 23800.0 26400.0 28700.0 30600.0 33000.0 46300.0 49000.0 53913.0 57400.0 60200.0 64300.0 68200.0 72600.0 77000.0 81800.0 85400.0 88000.0 91500.0 95200.0 99400.0 103300.0 106800.0 109800.0 112000.0 114500.0 117400.0 120400.0 123500.0 126900.0 129000.0 130600.0 132700.0 135100.0 137400.0 139900.0 141700.0 143300.0 144400.0 145600.0 147200.0 148700.0 150300.0 151700.0 152600.0 153400.0 154600.0 155700.0 156900.0 158000.0 159000.0 159900.0 160300.0 161200.0 162000.0 162800.0 163200.0 164100.0 164900.0 165200.0 165900.0 166400.0 167300.0 167800.0 168500.0 168900.0 169100.0 169600.0 170200.0 170700.0 171200.0 171600.0 171900.0 172200.0 172600.0 173100.0 173600.0 174100.0 174400.0 174700.0 174900.0 175300.0 175700.0 176300.0 176800.0 177100.0 177500.0 177700.0 178100.0 179100.0 179800.0 180300.0 181000.0 181300.0 181700.0 182200.0 182700.0 183100.0 183600.0 184000.0 184400.0 184600.0 185100.0 185500.0 186000.0 186400.0 186900.0 187200.0 187400.0 187800.0 188100.0 188600.0 189000.0 189400.0 189800.0 190000.0 190400.0 190800.0 191300.0 191800.0 192300.0 192700.0 192900.0 193500.0 194000.0 194600.0 195200.0 195900.0 196400.0 196800.0 197400.0 198100.0 198800.0 199500.0 200200.0 200800.0 201300.0 202100.0 203000.0 203900.0 204800.0 205800.0 206600.0 207100.0 208200.0 209300.0 210600.0 211900.0 213200.0 214200.0 214900.0 216200.0 217600.0 219100.0 220500.0 221900.0 222900.0 223700.0 225000.0 226500.0 228000.0 229400.0 230600.0 231400.0 232100.0 233300.0 234600.0 236000.0 237300.0 238700.0 239800.0 240700.0 242200.0 243700.0 245400.0 246900.0 248500.0 249700.0 250500.0 252500.0 254200.0 256000.0 257900.0 259500.0 260900.0 261900.0 263700.0 265600.0 267700.0 269600.0 271800.0 273500.0 274800.0 276900.0 279300.0 281900.0 284600.0 287600.0 290000.0 291900.0 294800.0 298300.0 302100.0 306100.0 310300.0 314100.0 317100.0 321600.0 326700.0 332800.0 339200.0 345700.0 351200.0 355900.0 363100.0 371500.0 381400.0 391600.0 402500.0 412000.0 419300.0 429600.0 441200.0 454800.0 467800.0 481700.0 493200.0 502300.0 515200.0 530200.0 546500.0 562700.0 579100.0 593100.0 603800.0 618800.0 636700.0 656400.0 676100.0 696100.0 711000.0 722300.0 739100.0 758800.0 779500.0 800000.0 820600.0 835700.0 846400.0 863300.0 881800.0 902100.0 922100.0 942100.0 957500.0 967900.0 984200.0 1003300.0 1025000.0 1047600.0 1069400.0 1085500.0 1097400.0 1115400.0 1136700.0 1160100.0 1184400.0 1184400.0 1223700.0 1236700.0 1255700.0 1277900.0 1302600.0 1328200.0 1328200.0 1368100.0 1381900.0 1401200.0 1424700.0 1451000.0 1474000.0 1494100.0 1511800.0 1525300.0 1545500.0 1570000.0 1596600.0 1620200.0 1641200.0 1657900.0 1672000.0 1691700.0 1716200.0 1741800.0 1762200.0 1780200.0 1795400.0 1807500.0 1823500.0 1844000.0 1866000.0 1883700.0 1898900.0 1911800.0 1921700.0 1935600.0 1954000.0 1973200.0 1991000.0 2008200.0 2020900.0 2029200.0 2041300.0 2057300.0 2073100.0 2087600.0 2101000.0 2112000.0 2119100.0 2128800.0 2141400.0 2154600.0 2165900.0 2176300.0 2185100.0 2190600.0 2198000.0 2207700.0 2217700.0 2226500.0 2235700.0 2243200.0 2248400.0 2255500.0 2264600.0 2274400.0 2283400.0 2292100.0 2299400.0 2304300.0 2310900.0 2319600.0 2328700.0 2337000.0 2345600.0 2352600.0 2358000.0 2365100.0 2374200.0 2383600.0 2392600.0 2401700.0 2409700.0 2415200.0 2423400.0 2433800.0 2445300.0 2456200.0 2467600.0 2477500.0 2484600.0 2494800.0 2507900.0 2521800.0 2535000.0 2548200.0 2560400.0 2569400.0 2581500.0 2597100.0 2614500.0 2631400.0 2647600.0 2661500.0 2671200.0 2683900.0 2700200.0 2718700.0 2736100.0 2752000.0 2765100.0 2775200.0 2787200.0 2803600.0 2824100.0 2845300.0 2865000.0 2882300.0 2893900.0 2910100.0 2931400.0 2954000.0 2975200.0 2995200.0 3012100.0 3024600.0 3040700.0 3061500.0 3084700.0 3107300.0 3128800.0 3147100.0 3159200.0 3175600.0 3196900.0 3220300.0 3240300.0 3259000.0 3275500.0 3286400.0 3300700.0 3320300.0 3340400.0 3358000.0 3374600.0 3387800.0 3397100.0 3408800.0];
endfunction
Anfängliches Infektionsgeschehen
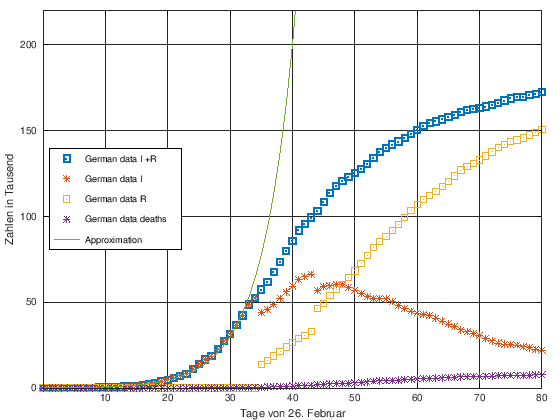
[Bearbeiten]Graphisch lässt sich das anfängliche Infektionsgeschehen wie folgt darstellen:
%aktuelle Daten: (passen nicht mehr zur exponenzieller Funktion)
A=coronaData();
inf_falleWHO=A(1,:);
inf_falleWHOrecovered=A(3,:);
inf_falleWHOtoten=A(2,:);
inf_falleWHOaktuell=inf_falleWHO-inf_falleWHOrecovered;
n=length(inf_falleWHO)
timesWHO=[0:1:n-1];
figure(1)
title ('Corona- infizierten Detuschland, Quelle RKI')
plot (timesWHO, inf_falleWHO./1000, 's', "linewidth",4,
timesWHO,inf_falleWHOaktuell/1000, '*' ,
timesWHO,inf_falleWHOrecovered/1000,'s', timesWHO,
inf_falleWHOtoten./1000 , '*' )
xlabel ('Tage von 26. Februar')
ylabel ('Zahlen in Tausend')
legend ( "German data I +R" ,"German data I ","German data R",
"German data deaths" ,"location", "west")
grid on
xticks([10,20,30,40,50,60,70,80])
axis([0,80, 0 ,220])
Man erkennt hierbei, dass sich bis zum 30. Tag die Gesamtanzahl der Infizierten durch eine passende Exponentialfunktion annähern lässt. Diese lässt sich durch den folgenden Befehl in Octave ergänzen:
y=0.1*e.^(0.19*timesWHO);
Gesamtes Infektionsgeschehen
[Bearbeiten]Das Gesamtgeschehen lässt sich analog zu vorherigem Abschnitt graphisch darstellen:
Um die Datenlage zu modellieren wird hier das SIR-Modell genutzt. Nach diesem wird die Bevölkerung in 3 Gruppen eingeteilt:
- Susceptible (Anfällige): Personen, welche mit der Krankheit angesteckt werden können.
- Infected (Infizierte): Personen, welche die Krankheit durchmachen und andere anstecken können.
- Removed (Entfernte): Personen, welche entweder immun sind (genesen) oder an der Krankheit verstarben.
Zwischen den Anzahlen der einzelnen Gruppen besteht folgender Zusammenhang:
Hierbei beschreibt eine zeitabhängige Infektionsrate (abhängig von den getroffenen Entscheidungen der Gesellschaft), ist die Wahrscheinlichkeit, dass eine infizierte Person als Infiziert erkannt wird (hier wurde zunächst von 10% ausgegangen), ist die maximal mögliche Anzahl an Infizierten (wurde hier als 2/3 der Gesamtbevölkerung geschätzt) und ist die Rate, mit der Menschen die Krankheit besiegen bzw. daran sterben.
Die Infektionsrate kann wie folgt aus den Daten bestimmt werden. Zum Vergleich wurde die Infektionsrate auch für den Fall bestimmt, dass die Zahl der Infizierten nicht beschränkt ist:

%infektionsrate aus den RKI-Daten berechnet: T=1 ccc(1)=cc(1); for i=T+1:n ccc(i-T)=(inf_falleWHO(i)-inf_falleWHO(i-T))/(inf_falleWHO(i- T)); %psi zum Zeitpunkt t=0.5 mit Diffusionskonstante 1 % Infektionsrate aus dem beschränktem Modell ccc2(i-T)=NM*(inf_falleWHO(i)-inf_falleWHO(i-T))/(inf_falleWHO(i-T)*(NM-0.001*inf_falleWHO(i-T))); endfor plot (ccc, '*', ccc2 , 's')
Hier lässt sich erkennen, dass sich die Infektionsrate in mehrere Teilintervalle gleichen Verhaltens einteilen lässt. Mit der Funktion "Slowdown" wird die Infektionsrate in diesen Bereichen approximiert.
function val=slowdown(x)
% Ab Tag t (Argument der Funktion) wir die Infektionsrate in mehreren Schritten sinken:
if x<29 val=1.1-x/29*0.2 ;
else if x<47 val=(-0.0097*x+0.5)/0.32450;
else if x<75 val=(-0.0012*x+0.12)/0.32450;
else if x<130 val=(0.0285+0.00036*x)/0.32450 ;
else if x<220 val=(0.085+x*0.00002)/0.32450;
else if x<250 val=(0.001*x-0.19)/0.32450;
val=3.15*val; % AH
else if x<275 val=(-0.0008*x+0.248)/0.32450;
val=3.15*val;
else if x<300 val=(3.1*0.033+(x-275)*0.0018)/0.32450;
else if x<350 val=(-0.00015*x+0.105)/0.32450;val=2.2*val;
else if x<380 val=3.3*(-0.00019*x+0.1+(x-350)*0.0015)/0.32450;
else val=14.3*0.016/0.32450+(x-380)*16.3*0.016/0.32450*0.025;
endif
endif
endif
endif
endif
endif
endif
endif
endif
endif
endfunction
Mit der Funktion "slowdown" kann nun wiederum die Infektionslage modelliert werden:

%Lösung des ODE Systems times=(0:0.1:1.3*n); %Anfangsbedingungen yo=[NM-16/1000;16/1000;0]; f=@(y,x) [-c*slowdown(x)*y(1)*y(2)/(r*NM);c*slowdown(x)*y(1)*y(2)/NM-w*y(2);w*y(2)-0*y(2)]; y = lsode (f, yo, times); figure(2) plot ( times, y (:,2), '-', times, y (:,3),'-',times, y (:,3)+y (:,2),'-') hold on plot (timesWHO, inf_falleWHO./1000, '.', "linewidth",4, timesWHO,inf_falleWHOaktuell/1000, '.' , timesWHO,inf_falleWHOrecovered/1000, '.' ) legend ( "Infected", "Removed", " Infected+Removed","German data I +R" ,"German data I ","German data R" ,"location", "west") axis([0,1.3*n, 0 ,3000]) hold on plot (timesWHO, inf_falleWHO./1000, '.', "linewidth",4, timesWHO,inf_falleWHOaktuell/1000, '.' , timesWHO,inf_falleWHOrecovered/1000, '.' ) legend ( "Infected", "Removed", " Infected+Removed","German data I +R" ,"German data I ","German data R" ,"location", "west")
Hier erkennt man, dass trotz guter Näherung der Infiziertenzahlen die modellierten Zahlen der Genesenen stark von den echten Daten abweichen. Zudem musste auch die Funktion "slowdown" weniger an die Daten der Infektionsrate als an die Infektionszahlen selbst angepasst werden. Außerdem fällt auf, dass bereits kleine Änderungen in der "slowdown"-Funktion zu großen Änderungen in den modellierten Infektionszahlen führen.
Wie folgt kann man sich den Fehler der Modellierung noch anzeigen lassen:

%relat. Fehler Infizierte %Interpolation der Funktionswerte an die RKI-Zeiten (Tage) I=interp1 (times, y(:,2), timesWHO+1); abs_I=abs(I-inf_falleWHOaktuell/1000); rel_I=abs(I-inf_falleWHOaktuell/1000)./(inf_falleWHOaktuell/1000); plot (timesWHO, rel_I) title ('relativer Fehler, Infected , Recovered') hold on %Fehlermaß maas_I=norm(abs_I)/norm(inf_falleWHOaktuell/1000) %weiterer Fehlermaß Durchschnitt und Euklid-Norm rel_I_mean=mean(rel_I) rel_I_norm=norm(rel_I); %relat. Fehler Genesene/Toten R=interp1 (times, y(:,3), timesWHO+1); abs_R=abs(R-inf_falleWHOrecovered/1000); rel_R=abs(R-inf_falleWHOrecovered/1000)./(inf_falleWHOrecovered/1000); plot (timesWHO, rel_R, '*') hold off legend('Infected', 'Recovered') %Fehlermaß maas_R=norm(abs_R)/norm(inf_falleWHOrecovered/1000) % weiterer Fehlermaß: Durchschnitt und Euklid-Norm rel_R_mean=mean(rel_R(36:length(timesWHO))) rel_R_norm=norm(rel_R(36:length(timesWHO)));
Berücksichtigung der Impfungen
[Bearbeiten]Ab dem 15.01.2021 wurde in Deutschland geimpft, somit hat sich ab dann die Anzahl der maximal möglichen Empfänglichen N verringert. Um dies zu beachten wurden zunächst die Daten des RKI bezüglich der täglich durchgeführten Zweitimpfungen wieder implementiert.
Impfungen=[ 0 0.610 0.906 17.362 15.771 34.964 51.584 41.907 35.559 44.959 23.147 38.685 48.439 59.638 46.098 57.646 52.167 30.533 61.486 67.732 95.445 70.776 77.174 53.065 26.096 56.783 74.221 76.798 67.443 76.095 45.579 25.265 55.632 54.694 55.776 51.296 54.841 37.115 29.107 60.108 59.759 59.583 58.507 65.425 40.029 24.285 49.384 53.698 69.059 62.836 67.409 49.305 34.462 52.505 54.742 65.745 60.894 71.717 49.439 35.993 58.894 67.036 79.509 75.353 82.847 53.862 38.762 76.189 83.513 88.191 80.002 88.656 63.177 52.750 89.034 93.359 103.115 92.615 71.658 66.012 53.518 73.375 99.281 97.184 89.794 88.768 65.551 50.020 76.911 73.605 83.524 71.035 75.368 56.518 39.188 60.850 61.886 71.919 66.335 67.946 59.851 47.866 73.340 90.484 127.377 132.811 137.762 76.382 67.960 116.008 162.431 227.883 219.748 231.902 154.720 103.519 192.554 284.650 362.851 159.407 230.327 164.932 122.007 204.786 355.842 533.059 487.112 436.746 258.427 159.531 181.350 377.132];
Anschließend wurde die maximale Anzahl der Empfänglichen dann für jeden einzelnen Tag berechnet:
NMV=ones(1, 456);
NMV=NMV*83000;
t=0
i=1
while i<n+1
t=i-324;
if i>325
NMV(i)=NMV(i)-sum(Impfungen(1:t));
else NMV(i)=NMV(i);
endif
i=i+1;
endwhile
NMV=NMV*2/3;
Betrachtet man die täglichen Impfungen, so fällt auf, dass sich mit gleitendem Durchschnitt dargestellt, auch hier wieder einzelne Bereiche gleichen Verhaltens finden lassen:
V=0*ones(1, n); V(n-n2+1:n)=V(n-n2+1:n)+Impfungen; %psi zum Zeitpunkt t=0.5 mit Diffusionskonstante 1 % 7- Tage Durchschnitt, moving Average for i=n-n2+1+4:n-3 Vaver(i)=sum(V(i-3:i+3))/7; endfor length(Vaver) function val=vaccin(x) if x<325 val=0.0 ; else if x<343 val=3.1097*(x-325); else if x<421 val= 3.1097*(343-325)+0.08*(x-343); else val= 3.0597*(343-325)+0.08*(421-343)+8.0*(x-421); endif endif endif endfunction % fuer graphische Darstellung for i=timesWHO+1 vacc_vektor(i)=vaccin(timesWHO(i)); endfor figure(2) plot(timesWHO, V) hold on plot (timesWHO, vacc_vektor) plot (timesWHO(1:n-3), Vaver) title('Vaccinated') axis([300,n,0,530]) legend('Geimpfte, Data', 'Geimpfte, Funktion' ,'Geimpfte Vaver', "location",'west') grid on
In folgendem Plot sieht man sowohl die echten Daten, als auch die Modellierung.

Anzumerken ist hier, dass nach langer Zeit mit der implementierten Funktion NMf negative Werte auftreten würden, was nicht realistisch wäre. Daher muss hierauf geachtet werden.
Baut man nun die Impfungen in das SIR-Modell ein, so kann dies wie folgt aussehen:
f=@(y,x) [-c*slowdown(x,25)*y(1)*y(2)/(r*NM)- vaccin(x, 0.158*NM);c*slowdown(x,25)*y(1)*y(2)/NM-w*y(2);w*y(2)-0*y(2)+ 1*vaccin(x,0.158*NM)];
yy = lsode (f, yo, times);
plot (timesWHO, inf_falleWHO./1000, '.', "linewidth",4, timesWHO,inf_falleWHOaktuell/1000, '.' , timesWHO,inf_falleWHOrecovered/1000, '.' )
plot ( times, yy (:,2),times, yy (:,1),"b" )
legend ("German data I +R" ,"German data I ","German data R" , "Infected mit vaccin", "Suspected mit vaccin", "location", "west")
Tutorium 4 - FDM für Poissongleichung
[Bearbeiten]In Aufgabenteil a) der Tutoriumsaufgabe IV sollte die Finite-Differenzen-Methode (FDM) für die Dirichlet-Randwertaufgabe mit inhomogenen Randbedingungen auf einem rechteckigen Gebiet implementiert werden.
Bei Aufgabenteil b) sollte die Finite-Differenzen-Methode (FDM) für die Neumann-Randwertaufgabe auf dem Rechteck D mit homogenen Randbedingungen mit implementiert werden.
Aufgabenteil a): Implementierung der Randwertaufgabe für Dirichlet-Randbedingungen auf dem Rechteck
[Bearbeiten]Die stationäre Diffusion wird mithilfe der Poissongleichung auf einem rechteckigen Gebiet und der Finiten Differenzen Methode implementiert:
Zuerst definieren wir die Quelldichtefunktion auf der rechten Seite der Poissongleichung.

function wert=Quellfunktion(x,y)
if sqrt((x.-3.25).^2+(y.-3).^2)<=0.35 wert=10;
else wert=0;
endif
endfunction
Nun wird das Gebiet durch die Endpunkte der Intervalle festgelegt. Die konstante Schrittweite wird durch die Anzahl der inneren X-Punkte bestimmt, wobei die Gitterpunkte wie folgt definiert sind: , mit
%definiere Gebiet D xend=8; yend=5; %Anzahl von X-Punkten, verkleinern Sie diese um die Struktur der Matrix in weiteren Zellen besser zu erkennen N=50 %-------------------- hx=xend/(N+1) hy=hx; M=floor((yend/hy))-1 %Koordinatenmatrix der Gitterpunkte [x,y]=meshgrid(hx:hx:xend-hx,hy:hy:yend-hy);
Als nächstes wird der konstante Diffusionskoeffizient festgelegt und die blocktridiagonale Systemmatrix implementiert. Die Einträge befinden sich dabei nur auf der Diagonalen, auf den beiden Nebendiagonalen und auf der N-ten und -N-ten Nebendiagonale.

%konstanter Diffusionskoeffizient a=1.0; %konstanter Diffusionskoeffizient %-------------------------- %RANDBEDINGUNGEN Vorbereitung fuer eine Teilaufgabe %------------------------------------------- %Dirichlet RB: V=[1:1:50]; wert_unten=0.01*V; wert_oben=-0.02*V; wert_links=-1/225*V.*V+2/15*V wert_rechts=+0.05*V-1; %-------------------------------------------
%Systemmatrix %------------------------------------------- Vec1=ones(N*M,1); %erzeugt Vektor aus Einsern der Laenge N*M
%Systemmatrix N^2xM^2 mit -4 auf der Diagonale und 1 auf den Nebendiagonalen und 1 auf den N und -N-ter Nebendiagonale BB=diag(-4.*Vec1,0)+diag(ones(N*M-1,1),1)+ diag(ones(N*M-1,1),-1)+diag(ones(N*(M-1),1),N)+diag(ones(N*(M-1),1),-N);
%Korrektur der Matrix (Blocktridiagonalitaet) for i=1:(M-1) BB(i*N+1,i*N)=0;BB(i*N,i*N+1)=0; endfor
%------------------------------------------- %Matrix mit Diffkoeff/h^2 multiplizieren %------------------------------------------- BB=BB*a/hx^2;
%Matrixdarstellung - optional %----------------------------- figure (1); %surface (BB) spy (BB); xlabel("Spalten") ylabel("Zeilen") zlabel("Matrix B") title ("Systematrix B");
Die Matrix der rechten Seite der Poissongleichung wird in jedem Gitterpunkt definiert, um die Matrix mit Beiträgen aus den Dirichlet-Randbedingungen zu erweitern. Die Matrix als Vektor spaltenweise umgeordnet.
%rechte Seite des Systems clear f; for i=1:N for j=1:M f(j,i)=1*Quellfunktion(x(j,i),y(j,i)); %Dirichlet Randbedingung unten und Oben: if j==1 f(j,i)=1*Quellfunktion(x(j,i),y(j,i))+ a*wert_unten(i)/hx^2; endif if j==M f(j,i)=1*Quellfunktion(x(j,i),y(j,i))+ a*wert_oben(i)/hx^2; endif %Dirichlet Randbedingung links und rechts: %...if (i==1)|| (i==N ) if i==1 f(j,i)=1*Quellfunktion(x(j,i),y(j,i))+a*wert_links(j)/hx^2; endif if i==N f(j,i)=1*Quellfunktion(x(j,i),y(j,i))+a*wert_rechts(M+1-j)/hx^2; endif %endif endfor endfor
% Wichtig!: Ecken-Beiträge: da in den Ecken jeweils der Differenzenquotient in x (Wert links oder rechts) %als auch in y Richtung (oben/unten) approximiert und aufaddiert werden muss %--------------------------------- f(1,1)=1*Quellfunktion(x(j,i),y(j,i))+ a*wert_links(M)/hx^2+ a*wert_unten(1)/hx^2; f(1,N)= 1*Quellfunktion(x(j,i),y(j,i))+ a*wert_rechts(M)/hx^2+ a*wert_unten(N)/hx^2; %f(1,1); f(M,1)=1*Quellfunktion(x(j,i),y(j,i))+ a*wert_links(1)/hx^2+ a*wert_oben(1)/hx^2; f(M,N)=1*Quellfunktion(x(j,i),y(j,i))+ a*wert_rechts(1)/hx^2+ a*wert_oben(N)/hx^2; %f(M,1);
size (f)
%reshape funktioniert Spaltenmässig: es wird Spalte1, dann Spalte2, usw eingeordnet, wobei wir die Matrix f nach Reihen/Zeilen einordnen. %Daher soll die Matrix f' mit N(Zeilen)x M(Spalten) in ein langern Vektor eingeordnet. b=-1*reshape(f',N*M,1);
Mit dem Befehl 'x=A\b' wird die Lösung eines linearen Systems gefunden. Auf diese Weise wird unser Gleichungssystem gelöst. Der erhaltene Lösungsvektor wird dann wieder zurück in eine Lösungsmatrix überführt. Da die Matrix N Zeilen und M Spalten hat, werden die inneren Gitterpunkte erst nach dem Transponieren durch die Lösungsapproximationsmatrix repräsentiert.


%LOESUNGSSCHRITT %------------------------------------------------- sol=BB\b;
sol_matrix=reshape(sol,N,M);% Matrix mit N(Zeilen)x M(Spalten) sol_matrix=sol_matrix'; % Transponiert, wegen reshape %-------------------------------------------------
%Oeffne ein Bildfenster und plotte die Lösung auf dem realen Koordinatenfeld %------------------------------------------------- figure(2); surfc(x,y,sol_matrix); title ("Loesung"); ylabel("y") xlabel("x") colormap("hsv") colorbar
figure(3); surfc(x,y,sol_matrix); title ("Loesung"); ylabel("y") xlabel("x") colormap("hsv") colorbar view([0.5,0.5,0.5])
%weiteres Bild mit Quellfunktion- rechte Seite des Systems figure(4); surfc(x,y,f); title ("Quellfunktion"); ylabel("y") xlabel("x") %colormap("hsv")
figure(5); surfc(x,y,f); title ("Quellfunktion"); ylabel("y") xlabel("x") view([0.5,0.5,0.5])
Aufgabenteil b): Implementierung der Randwertaufgabe für Neumann-Randbedingungen auf dem Rechteck
[Bearbeiten]Zuerst definieren wir ein Rechteck. Die Blocktridiagonalmatrix wird um neue Blockzeilen und Blockspalten erweitert . Dabei ist jeder Block dieser Matrix eine Matrix . N wird mit N+2 und M mit M+2 ersetzt. Das bedeutet, dass auf der Diagonalen ein Vektor der Länge gesetzt wird. Zur Implementierung der approximierten Richtungsableitungen an den Rändern sollen die ersten zwei Blöcke der ersten Blockzeile und die letzten zwei Blöcke der letzten Blockzeile sowie jeweils die erste und letzte Zeile der einzelnen Blöcke ersetzt werden. Um eine Lösung zu erhalten werden drei Randbedingungen durch Neumann und die Bedingung für den unteren Rand durch Dirichlet definiert.

[x,y]=meshgrid(hx-hx:hx:xend,hy-hy:hy:yend);
N=N+2 M=M+2
%konstanter Diffusionskoeffizient a=1.0; %konstanter Diffusionskoeffizient %-------------------------- %RANDBEDINGUNGEN Vorbereitung fuer eine Teilaufgabe %------------------------------------------- %Dirichlet RB: wert_unten=5;
%-------------------------------------------
%Systemmatrix %------------------------------------------- Vec1=ones(N*M,1); %erzeugt Vektor aus Einsern der Laenge N*M
%Systemmatrix N^2xM^2 mit -4 auf der Diagonale und 1 auf den Nebendiagonalen und 1 auf den N und -N-ter Nebendiagonale BB=diag(-4.*Vec1,0)+diag(ones(N*M-1,1),1)+ diag(ones(N*M-1,1),-1)+diag(ones(N*(M-1),1),N)+diag(ones(N*(M-1),1),-N);
%Korrektur der Matrix (Blocktridiagonalitaet) for i=1:(M-1) BB(i*N+1,i*N)=0;BB(i*N,i*N+1)=0; endfor
%------------------------------------------- %Matrix mit Diffkoeff/h^2 multiplizieren %------------------------------------------- BB=BB*a/hx^2;
%Matrixdarstellung - optional %----------------------------- figure (6); %surface (BB); spy (BB); xlabel("Spalten") ylabel("Zeilen") zlabel("Matrix B") title ("Systematrix BB");
%Randbedingung Neumann c=a h=hx block=diag((c/h)*ones(N,1)); %oberer Rand BB(N*(M-1)+1:N*M,N*(M-1)+1:N*M)=block; BB(N*(M-1)+1:N*M,N*(M-2)+1:N*(M-1))=-block; %-------------------------- %Neumann RB für x %---------------------------------------------------------------------- for i=1:(M-2) vector_up=zeros(1,N*M); vector_up(N*i+1)=c/h; vector_up(N*i+2)=-c/h; vector_down=zeros(1,N*M); vector_down(N*i+N)=c/h; vector_down(N*i+N-1)=-c/h; BB(i*N+1,:)=vector_up ; BB(i*N+N,:)=vector_down ; endfor
BB(1:N,1:N);
Im Folgenden wird die rechte Seite der Poissongleichung definiert. Ebenso wie in Aufgabenteil a) wird zum Lösen des Gleichungssystems die Matrix in einen Vektor überführt und anschließend wieder zu einer Lösungsapproximationsmatrix transponiert. Dann wird unsere Quellfunktion und die erhaltene Lösung geplottet.
%rechte Seite des Systems clear f; for i=1:N for j=1:M f(j,i)=1*Quellfunktion(x(j,i),y(j,i)); %Dirichlet Randbedingung unten und Oben: if j==1 f(j,i)=1*Quellfunktion(x(j,i),y(j,i))+ a*wert_unten/hx^2; endif
% ausserdem muessen noch die untere Eckpunkte um Dirichlet Werte ergaenzt werden und zwar 2-fach... f(1,1)=1*Quellfunktion(x(1,1),y(1,1))+ 2*a*wert_unten/hx^2; f(1,N)=1*Quellfunktion(x(1,N),y(1,N))+ 2*a*wert_unten/hx^2; endfor endfor
size (f)
%reshape funktioniert Spaltenmässig: es wird Spalte1, dann Spalte2, usw eingeordnet, wobei wir die Matrix f nach Reihen/Zeilen einordnen. %Daher soll die Matrix f' mit N(Zeilen)x M(Spalten) in ein langern Vektor eingeordnet. b=-1*reshape(f',N*M,1);



%LOESUNGSSCHRITT %------------------------------------------------- sol=BB\b;
sol_matrix=reshape(sol,N,M);% Matrix mit N(Zeilen)x M(Spalten) sol_matrix=sol_matrix'; % Transponiert, wegen reshape %-------------------------------------------------
%Oeffne ein Bildfenster und plotte die Lösung auf dem realen Koordinatenfeld %------------------------------------------------- figure(7); surfc(x,y,sol_matrix); title ("Loesung Neumann RB + Dirichlet RB unten"); ylabel("y") xlabel("x") colormap("hsv") colorbar
figure(8); surfc(x,y,sol_matrix); title ("Loesung Neumann RB + Dirichlet RB unten"); ylabel("y") xlabel("x") colormap("hsv") colorbar view([0,0.5,0.5])
%weiteres Bild mit Quellfunktion- rechte Seite des Systems figure(9); surfc(x,y,f); title ("Quellfunktion"); ylabel("y") xlabel("x") %colormap("hsv")
figure(10); surfc(x,y,f); title ("Quellfunktion"); ylabel("y") xlabel("x") view([0.5,0.5,0.5])
Tutorium 5 - Epidemiologische Modellierung als Reaktionsdiffusionsprozess mit FDM
[Bearbeiten]Ergänzungen Octave-Tutorial
[Bearbeiten]Diese Ergänzungen wurden bei dem Octave-Tutorial vorgenommen, um die Implementation der Gruppe bzgl. der verwendeten Octave-Befehle nachvollziehbar zu machen.
- Matrix-Multiplikation in Octave - notwendig für die Modellierung diskreter Transportprozesse.
Skripte in Octave
[Bearbeiten]Referenzieren Sie hier die von Ihnen verwendeten Skripte und Bibliotheken in Octave oder R/Studio
Literatur
[Bearbeiten]Notieren Sie hier die von Ihnen verwendete Literatur
- Quelle 1