Kurs:Räumliche Modellbildung/Gruppe 3
Gruppenseite - D
[Bearbeiten]Diese Seite enthält die gesammelten Materialien der Gruppe 3 - D für die Portfolioprüfung.
Teilnehmer-innen
[Bearbeiten]- Dilg, Anna-Katharina
Aufgabe Tutorium
[Bearbeiten]Ziel
[Bearbeiten]Darstellung des Verlaufs von COVID-19innerhalb von 210 Tagen in jedem Bundesland Deutschlands anhand eines SIR-Modells. Die nachfolgenden Skripte wurden in Octave erstellt.
Die Modellierung umfasst drei Zyklen, welche aufeinander aufbauen. Der erste Zyklus bildet somit die Basis für die beiden darauffolgenden Zyklen. Es sollen die Auswirkungen von Reisen zwischen Bundesländern, sowie einschränkende Maßnahmen wie die Maskenpflicht oder Schulschließungen graphisch dargestellt werden. Die Plots der COVID-19 Ausbreitung je Bundesland sollen Aufschluss darüber geben, welche Faktoren maßgeblich für eine erhöhte Infiziertenanzahl (infected) sind:
- hat die Einreise aus anderen Bundesländern großen Einfluss?
- müssen einige Bundesländer früher interne Maßnahmen einleiten?
- wie stark sind die Auswirkungen der eingeleiteten Maßnahmen?
- ...
Im Folgenden werden die Zyklen nun nacheinander vorgestellt und erläutert.
Zyklus 1
[Bearbeiten]Jeder Zyklus besteht aus einem Hauptskript, welches zum einen den dazugehörigen epidemiologischen Prozess und zum anderen den Transportprozess aufruft. Das Hauptskript führt alle Teile zusammen und plottet am Ende die Funktionen aller Bundesländer. Das Ziel ist es hier ein einfaches SIR-Modell aufzustellen (mit Removed), welches den Verlauf von COVID-19 ohne(!) Lockdown oder anderweitige Maßnahmen darstellen soll. Im ersten Zyklus wird es nur eine Transportmatrix, sowie eine feste Infektionsrate je Bundesland geben. Dies bedeutet das angenommen wird, dass es von Seiten der Bundesländer aus in diesem Verlauf keine Maßnahmen bezüglich der Pandemie gibt (Reisebeschränkungen, Hygienemaßnahmen, ...). Es wird außerdem davon ausgegangen, dass man sich höchstens 1 Mal infizieren kann. Dies entspricht jedoch nicht der Realität.
Hauptskript
[Bearbeiten]Im ersten Teil des Hauptskriptes wird zunächst die Zeit festgelegt, in welcher der Verlauf betrachtet wird. Hier wurde Tage gewählt. Durch den Befehl linspace wird hier ein Zeilenvektor mit äquidistanten Werten von bis erstellt.
N_t=210
% X-Achseneinteilung
t=linspace(-2, N_t, N_t+1);%t ist Vektor
Anzahl_Bundeslaender=16;
Im ersten Teil des Hauptskriptes wird nun der epidemiologische Prozess aufgerufen. Dieser ausgelagerte Skriptteil wurde als Zyklus1_wikiversity_Epi (siehe weiter unten) bezeichnet und wird durch die Runden Klammern aufgerufen. Wichtig ist hierbei, dass die Skripte im gleichen Verzeichnis gespeichert wurden. Als nächstes muss festgelegt werden, wie die Anfälligen (Susceptible), die Infizierten (Infected), die Genesenen (Recovered) und die Toten (Removed) zusammenhängen:
Zyklus1_wikiversity_Epi(); %die Funktion wird hier aufgerufen
Im Folgenden wird der Transportprozess aufgerufen durch die Funktion Zyklus1_wikiversity_Trans (Näheres siehe weiter unten).
Zyklus1_wikiversity_Trans(); %die Funktion wird hier aufgerufen
Epidemiologischer Prozess
[Bearbeiten]Hier wird das Skript Zyklus1_wikiversity_Epi näher beschrieben. Neben Infektions-, Genesungs- und Sterberate für jedes Bundesland werden hier die Vektoren der Susceptible, Infected, Recovered und Removed erzeugt.
% Infektionsrate (Daten hier nur abgeschätzt)
% SL RLP HS BW BY S TH NI SA BB BE HH BH MV SH NRW
beta=[0.28700 0.28000 0.29070 0.29450 0.29599 0.29000 0.27950 0.28070 0.27450 0.27201 0.30850 0.30450 0.29999 0.25450 0.28090 0.29310];
% Genesungsrate (an Tag 15 nicht mehr infiziert)
gamma=1/14;
% Sterberate (Daten hier nur abgeschätzt)
% SL RLP HS BW BY S TH NI SA BB BE HH BH MV SH NRW
delta=[0.0026 0.0030 0.0029 0.0032 0.0034 0.0030 0.0031 0.0029 0.0031 0.0036 0.0032 0.0032 0.0029 0.0031 0.0030 0.0028];
Anschließend wird festgesetzt, dass beispielsweise die Recovered den Wert im Zeitpunkt haben, da zu Beginn niemand die Infektion in irgendeinem Stadium durchläuft. An dieser Stelle wurde verbessert, dass die Sucseptible anfangs den Wert haben, da es ja schon Krankheitsträger (Indexpatienten) gibt. Daher ergibt sich eigentlich je Bundesland . Allerdings ist die Anzahl an Krankheitsträgern so gering, dass dieser Fehler nicht stärker ins Gewicht fallen würde.
% Vektoren erstellen
S=zeros(N_t+1, Anzahl_Bundeslaender); % Vektor nur mit Nullen
I=zeros(N_t+1, Anzahl_Bundeslaender);
R=zeros(N_t+1, Anzahl_Bundeslaender);
Rem=zeros(N_t+1, Anzahl_Bundeslaender);
% Vektoren Susceptible, Infected, Recovered je Bundesland (Werte zu Beginn für alle gleich)
for i=1:Anzahl_Bundeslaender
S(1,i)=1-(1.93E-07); %Susceptible zu Beginn, S(1,1) hier dem Saarland zugordnet, S(1,16) wäre NRW
I(1,i)=1.93E-07; %Infected
R(1,i)=0; %Recovered
Rem(1,i)=0; %Removed
endfor
Der folgende Schritt ist besonders wichtig: Hier wird festgelegt, wie die einzelnen Stadien des Pandemieverlaufs zusammenhängen (von anfällig bis genesen/verstorben). Dabei wird immer von einem Tag aus dem Zeitintervall ausgegangen und der nächste Tag wird berechnet. Es handelt sich hierbei um ein diskretes Modell, d.h. es werden nur ganze Tage betrachtet und es kann nicht auf jeden Zeitpunkt dazwischen zugegriffen werden im Gegensatz zum kontinuierlichen Modell. Die Susceptible von morgen () bestehen beispielsweise aus den Susceptible von heute (also ), von denen eine Gruppe Menschen abgezogen wird, welche an Tag in den Zustand der Infected wechselt. Da lediglich auf Grund von Bewegung neue Anfällige in ein Bundesland gelangen können und nicht durch den epidemiologischen Prozess, so nimmt diese Zahl im Verlauf immer weiter ab. Susceptible kann man nämlich nicht werden. Entweder man hat sich bis zum Ende des Zeitintervalls nie infiziert, oder man hat den Zustand gewechselt. Die finalen Zustände sind daher Susceptible, Recovered oder Removed, da hier ebenfalls davon ausgegangen wird, dass es keine erneute Ansteckun nach einmaligem durchlaufen der Krankheit gibt.
% Zeitschritte pro Tag
for n=1:N_t %Zeit läuft von 1 Tag bis N_t
%zunächst Berechnung jedes Bundesland
for i=1:Anzahl_Bundeslaender
S(n+1,i)=S(n,i)-beta(1,i)*S(n,i).*I(n,i); %Infizierte werden abgezogen
I(n+1,i)=I(n,i)+beta(1,i)*S(n,i).*I(n,i)-gamma.*I(n,i); %neue Infizierte kommen hinzu, Recovered werden abgezogen
R(n+1,i)=R(n,i)+gamma.*I(n,i)-delta(1,i)*I(n,i); %neue Recovered kommen hinzu, Removed werden abgezogen
Rem(n+1,i)=Rem(n,i)+delta(1,i)*I(n,i); %Tote von zuvor plus Infizierte mal Sterberate (neue Removed)
endfor
endfor
Transportprozess
[Bearbeiten]Der Transportprozess, gespeichert in Zyklus1_wikiversity_Trans, enthält die Transportmatrix. Diese gibt an, mit welcher Wahrscheinlichkeit Personen in einem Bundesland in ein anderes Reisen und je nach Lesart auch umgekehrt. Diese Werte sind nicht symmetrisch angeordnet, da beispielsweise Haushalte umziehen, längere geschäftliche Aufenthalte tätigen o.Ä.. Jedoch gibt es eine Orientierung an verschiedenen Pendlerkarten um den Bezug zur Realität besser darstellen zu können. Verbindendes Element in allen Bundesländern die Hauptdiagonale mit vergleichsweise hohen Werten, da davon auszugehen ist, dass der Großteil der Bevölkerung eines Bundeslandes vor Ort bleibt. Es ist ebenfalls anzumerken, dass es sich um ein geschlossenes System handelt (Summe jeder Spalte ist 1). Bezogen auf dieses Modell heißt dies, dass ein Austausch der Population über die Grenzen Deutschlands hinweg ausgeschlossen ist. Es kommen somit keine Menschen (Susceptible,Recovered oder Removed über die Grenzen nach Deutschland oder verlassen das Land. Der Austausch ist auf die 16 Bundesländer untereinander beschränkt.
%Austauschmatrix:
%Dimension = Anzahl_Bundeslaender
% SL RLP HS BW BY S TH NI SA BB BE HH BH MV SH NRW
A=[0.80, 0.03, 0.01, 0.01, 0.00, 0.00, 0.00, 0.01, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.01; % SL %Hoher Austausch mit RLP
0.12, 0.86, 0.05, 0.05, 0.02, 0.03, 0.01, 0.01, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.04; % RLP
0.01, 0.01, 0.83, 0.01, 0.01, 0.03, 0.04, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.01; % HS
0.04, 0.03, 0.01, 0.76, 0.04, 0.00, 0.01, 0.01, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.01; % BW %sehr viele Pendler in BW nach außerhalb
0.01, 0.01, 0.03, 0.10, 0.91, 0.01, 0.01, 0.00, 0.00, 0.00, 0.04, 0.00, 0.00, 0.00, 0.00, 0.01; % BY
0.00, 0.00, 0.01, 0.01, 0.00, 0.77, 0.02, 0.00, 0.01, 0.01, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00; % S
0.00, 0.01, 0.03, 0.03, 0.01, 0.08, 0.79, 0.00, 0.01, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00; % TH
0.00, 0.00, 0.00, 0.01, 0.00, 0.00, 0.01, 0.73, 0.00, 0.00, 0.00, 0.03, 0.02, 0.00, 0.01, 0.01; % NI
0.00, 0.00, 0.01, 0.00, 0.00, 0.02, 0.02, 0.00, 0.91, 0.01, 0.01, 0.00, 0.00, 0.00, 0.00, 0.00; % SA
0.00, 0.00, 0.00, 0.00, 0.00, 0.01, 0.05, 0.02, 0.02, 0.85, 0.01, 0.00, 0.00, 0.01, 0.00, 0.00; % BB
0.01, 0.01, 0.01, 0.01, 0.00, 0.03, 0.00, 0.00, 0.02, 0.11, 0.90, 0.00, 0.00, 0.01, 0.02, 0.00; % BE %Die meisten Einreisenden kommen aus Brandenburg
0.00, 0.01, 0.00, 0.00, 0.01, 0.00, 0.01, 0.10, 0.00, 0.00, 0.01, 0.91, 0.03, 0.03, 0.00, 0.01; % HH
0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.01, 0.06, 0.00, 0.00, 0.00, 0.02, 0.92, 0.01, 0.01, 0.00; % BH
0.00, 0.00, 0.00, 0.00, 0.00, 0.02, 0.01, 0.02, 0.03, 0.01, 0.02, 0.00, 0.01, 0.90, 0.01, 0.00; % MV
0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.01, 0.01, 0.00, 0.01, 0.01, 0.04, 0.01, 0.04, 0.94, 0.00; % SH
0.01, 0.03, 0.01, 0.01, 0.00, 0.00, 0.00, 0.03, 0.00, 0.00, 0.00, 0.00, 0.01, 0.00, 0.01, 0.90]; % NRW
%An sich geschlossenes System (Spalten = 1), das heißt keiner verlässt diese Bundesländer, außer durch Tod (Removed)
Nun muss noch der Bevölkerungsvektor angegeben werden. Dies entspricht jeweils den Anfälligen pro Bundesland zum Zeitpunkt (Ursprungspopulation) in Tausend.
%Populationen der Bundesländer zum Zeitpunkt 0 in Tausend
% SL RLP HS BW BY S TH NI SA BB BE HH BH MV SH NRW
b_0=[990, 4085, 6266, 11070, 13080, 4078, 2137, 7982, 2280, 2520, 3769, 1899, 683, 1610, 2890, 17930];
Nun muss zuerst der Bevölkerungsvektor verrechnet werden, um anschließend mit Hilfe der Austauschmatrix eine Bewegung der Population der einzelnen Bundesländer untereinander zu generieren.
Sneu=round(S.*b_0); %Bevölkerung je Bundesland wird mit einbezogen
Ineu=round(I.*b_0);
Rneu=round(R.*b_0);
Remneu=round(Rem.*b_0);
q=1:Anzahl_Bundeslaender;
% Zeitschritte pro Tag
for n=1:N_t
%Austauch zwischen den Bundesländern, siehe Austauschmatrix A
Sneu1(n+1,q)=Sneu(n+1,q)*A;
Ineu1(n+1,q)=Ineu(n+1,q)*A;
Rneu1(n+1,q)=Rneu(n+1,q)*A;
Remneu1(n+1,q)=Remneu(n+1,q)*A;
endfor
Plot
[Bearbeiten]Hier wird Beispielhaft ein Plot gezeigt, welcher für alle weiteren Zyklen in leicht angepasster Form verwendet werden kann. Das Speichern der Bilder ist optional, ist jedoch zu empfehlen.
%%%-------------- Teil 3: Plotten ------------------------------------------------------------------------------------------------
% Plot
for j=1:Anzahl_Bundeslaender %Schleife liefert 16 Bilder mit Plot je Bundesland
figure (j);
%Plotten
plot(t,Sneu1(:,j),'g',t,Ineu1(:,j),'r',t,Rneu1(:,j),'y',t,Remneu1(:,j),'k');
grid on
% Achseneinteilung
axis([0 N_t 0 b_0(1,j)+1200]) %Achse an Bevölerung angepasst
% Legende
legend('Susceptible','Infected','Recovered','Removed');
xlabel('Tage');
ylabel('Bevölkerung in Tausend');
set(gca,'XTick',0:15:N_t);
endfor
figure (1)
title('Saarland')
figure (2)
title ('Rheinland-Pfalz')
figure (3)
title ('Hessen')
figure (4)
title ('Baden-Württemberg')
figure (5)
title ('Bayern')
figure (6)
title('Sachsen')
figure (7)
title ('Thüringen')
figure (8)
title ('Niedersachsen')
figure (9)
title ('Sachsen-Anhalt')
figure (10)
title ('Brandenburg')
figure (11)
title('Berlin')
figure (12)
title ('Hamburg')
figure (13)
title ('Bremen')
figure (14)
title ('Mecklenburg-Vorpommern')
figure (15)
title ('Schleswig-Hollstein')
figure (16)
title('Nordrhein-Westfahlen')
% Speichern der Bilder
for j=1:Anzahl_Bundeslaender
%Speichern der Bilder
a03=["Zyklus 1, Bundesland Nr._", num2str(j),".jpg"]
saveas(j, a03)
endfor
Ergebnis/Interpretation
[Bearbeiten]Wie hier deutlich zu erkennen ist, ist die Infektionskurve Mecklenburg-Vorpommerns (links) deutlich flacher und erreicht ihr Maximum später im Vergleich zu anderen Bundesländern. Hamburg und Schleswig-Hollstein werden in diesem Fall genauer untersucht, da Einwohner Mecklenburg-Vorpommerns gerne nach Schleswig-Hollstein und Hamburg reisen und diese Gebiete offensichtlich ein erhöhtes Infektionsrisiko bieten. In diesem Fall wäre es ratsamer - sofern möglich - im eigenen Bundesland zu verweilen.
Ein Austausch zwischen Bayern, Hessen und Berlin sollte ebenfalls reduziert werden, da diese drei Bundesländer im Vergleich zu anderen einen kritischeren Verlauf der Pandemie haben, auch wenn dieser Unterschied hier nur minimal erkennbar ist.
An sich ist anzumerken, dass der Pandemieverlauf in den Bundesländern insgesamt recht ähnlich verläuft. Dies wahrscheinlich darauf zurückzuführen, dass von keinerlei Beschränkungen/Maßnahmen ausgegangen wird und dies sowohl für den epidemiologischen als auch den Transportprozess gilt. Dies muss im Folgenden näher untersucht werden.
Optimierung
[Bearbeiten]Im weiteren Verlauf der Modellierung kann die Wechselrate zu den Genesenen angepasst werden, da hier noch von einer Genesung (oder Tod) nach 14 Tagen ausgegangen wird. Durch Zufallszahlen kann man sich hier der Realität annähern. Zudem können durch weitere Transportmatrizen Ein- bzw. Ausreisebeschränkungen generiert werden, welche in bestimmten Zeitfenstern individuell für Bundesländer gelten. Ebenso können diese Zeitintervalle auf eine variierende Infektionsrate angewandt werden, welche Kontaktverbote, Maskenpflicht u.Ä. widerspiegeln sollen.
Zyklus 2
[Bearbeiten]Ziel
[Bearbeiten]In Zyklus 2 wird davon ausgegangen, dass es individuelle Maßnahmen seitens der Bundesländer gibt. Im
Hauptskript
[Bearbeiten]Hier werden für Maßnahmen (siehe unten) der Bundesländer bestimmte Zeitpunkte festgelegt, woraus sich verschiedene Zeitintervalle ergeben wie .
N_t1=50
N_t2=80
N_t3=100
Da der epidemiologische Prozess angepasst wurde (siehe unten), muss ein anderes Skript an dieser Stelle aufgerufen werden als zuvor.
Zyklus2_wikiversity_Epi();
Und auch der Austausch zwischen den Bundesländern ändert sich (siehe unten).
Zyklus2_wikiversity_Trans();
Epidemiologischer Prozess
[Bearbeiten]Die Infektionsrate ist im Zyklus 2 an den Verlauf der Pandemie angepasst. Das bedeutet, dass in einigen Bundesländern Hygienemaßnahmen (oder anderes) in Kraft tritt, um zunächst die Infektionsrate zu senken. In einigen Bundesländern geschieht dies früher als in anderen.
% Infektionsrate (Daten hier nur abgeschätzt)
for n=1:N_t
if n<N_t1
% SL RLP HS BW BY S TH NI SA BB BE HH BH MV SH NRW
beta=[0.28700 0.28000 0.29070 0.29450 0.29599 0.29000 0.27950 0.28070 0.27450 0.27201 0.30850 0.30450 0.29999 0.25450 0.28090 0.29310];
elseif n<N_t2 %einige Bundesländer nehmen leichte Einschränkungen vor
% SL RLP HS BW BY S TH NI SA BB BE HH BH MV SH NRW
beta=[0.25705 0.25609 0.27000 0.28250 0.29599 0.28900 0.27000 0.27100 0.26450 0.27001 0.30250 0.29300 0.29000 0.24700 0.27990 0.27990];
elseif n<N_t3 %stärkere Maßnahmen für ALLE Bundesländer, Einschränkungen HH erst sehr spät
% SL RLP HS BW BY S TH NI SA BB BE HH BH MV SH NRW
beta=[0.21400 0.22900 0.25010 0.25900 0.24090 0.26000 0.25050 0.26070 0.25700 0.25001 0.28050 0.28050 0.27370 0.23500 0.27090 0.24010];
else % vieler Orts leichte Lockerungen bis hin zum "normalen" Alltag
% SL RLP HS BW BY S TH NI SA BB BE HH BH MV SH NRW
beta=[0.21400 0.23708 0.26070 0.29450 0.27599 0.27000 0.27950 0.27880 0.27120 0.26099 0.29850 0.28450 0.28360 0.24700 0.27770 0.29310];
endif
endfor
Die Wechselrate zu den Genesenen (Recovered) wurde hier verbessert und durch eine Zufallszahl ersetzt. Somit tritt die Genesung nicht immer genau nach 14 Tagen ein.
% Genesungsrate (an Tag 15 nicht mehr infiziert)
%Verbesserungsvorschlag: Zufallszahl zwischen 12 und 16
a = 12;
b = 16;
r = a+(b-a)*rand(1,1) %Befehl rand generiert Zufallszahl mit vier Nachkommastellen zwischen 0 und 1, rand(5,5) würde 5x5 Matrix mit diesen Zahlen erzeugen
for i=1:Anzahl_Bundeslaender
gamma(1,i)=[1/r];
endfor
Transportprozess
[Bearbeiten]Es gibt ebenfalls zwei neue Transportmatrizen, welche je nach Zeit den Austausch unter den Bundesländern verringert (nicht bei allen).
% SL RLP HS BW BY S TH NI SA BB BE HH BH MV SH NRW
B=[0.84, 0.03, 0.01, 0.01, 0.00, 0.00, 0.00, 0.01, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.01; % SL
0.10, 0.89, 0.05, 0.05, 0.02, 0.03, 0.01, 0.01, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.04; % RLP
0.01, 0.01, 0.84, 0.01, 0.01, 0.03, 0.04, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.01; % HS
0.02, 0.01, 0.01, 0.83, 0.04, 0.00, 0.01, 0.01, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.01; % BW
0.01, 0.01, 0.02, 0.04, 0.91, 0.01, 0.01, 0.00, 0.00, 0.00, 0.02, 0.00, 0.00, 0.00, 0.00, 0.01; % BY
0.00, 0.00, 0.01, 0.01, 0.00, 0.84, 0.02, 0.00, 0.01, 0.01, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00; % S
0.00, 0.01, 0.02, 0.02, 0.01, 0.03, 0.79, 0.00, 0.01, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00; % TH
0.00, 0.00, 0.00, 0.01, 0.00, 0.00, 0.01, 0.78, 0.00, 0.00, 0.00, 0.03, 0.02, 0.00, 0.01, 0.01; % NI
0.00, 0.00, 0.01, 0.00, 0.00, 0.02, 0.02, 0.00, 0.91, 0.01, 0.01, 0.00, 0.00, 0.00, 0.00, 0.00; % SA
0.00, 0.00, 0.00, 0.00, 0.00, 0.01, 0.05, 0.02, 0.02, 0.92, 0.01, 0.00, 0.00, 0.01, 0.00, 0.00; % BB
0.01, 0.01, 0.01, 0.01, 0.00, 0.01, 0.00, 0.00, 0.02, 0.04, 0.92, 0.00, 0.00, 0.01, 0.02, 0.00; % BE
0.00, 0.01, 0.00, 0.00, 0.01, 0.00, 0.01, 0.05, 0.00, 0.00, 0.01, 0.91, 0.01, 0.01, 0.00, 0.01; % HH
0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.01, 0.06, 0.00, 0.00, 0.00, 0.02, 0.94, 0.01, 0.01, 0.00; % BH
0.00, 0.00, 0.00, 0.00, 0.00, 0.02, 0.01, 0.02, 0.03, 0.01, 0.02, 0.00, 0.01, 0.94, 0.01, 0.00; % MV
0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.01, 0.01, 0.00, 0.01, 0.01, 0.04, 0.01, 0.02, 0.94, 0.00; % SH
0.01, 0.02, 0.01, 0.01, 0.00, 0.00, 0.00, 0.03, 0.00, 0.00, 0.00, 0.00, 0.01, 0.00, 0.01, 0.90]; % NRW
% SL RLP HS BW BY S TH NI SA BB BE HH BH MV SH NRW
C=[0.92, 0.01, 0.01, 0.01, 0.00, 0.00, 0.00, 0.01, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.01; % SL
0.04, 0.93, 0.02, 0.02, 0.01, 0.01, 0.01, 0.01, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.02; % RLP
0.01, 0.01, 0.90, 0.01, 0.01, 0.01, 0.02, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.01; % HS
0.01, 0.01, 0.01, 0.89, 0.02, 0.00, 0.01, 0.01, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.01; % BW
0.01, 0.01, 0.01, 0.01, 0.94, 0.01, 0.01, 0.00, 0.00, 0.00, 0.01, 0.00, 0.00, 0.00, 0.00, 0.01; % BY
0.00, 0.00, 0.01, 0.01, 0.00, 0.92, 0.02, 0.00, 0.01, 0.01, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00; % S
0.00, 0.01, 0.01, 0.02, 0.01, 0.01, 0.86, 0.00, 0.01, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.00; % TH
0.00, 0.00, 0.00, 0.01, 0.00, 0.00, 0.01, 0.87, 0.00, 0.00, 0.00, 0.01, 0.01, 0.00, 0.01, 0.01; % NI
0.00, 0.00, 0.01, 0.00, 0.00, 0.01, 0.01, 0.00, 0.93, 0.01, 0.01, 0.00, 0.00, 0.00, 0.00, 0.00; % SA
0.00, 0.00, 0.00, 0.00, 0.00, 0.01, 0.02, 0.02, 0.02, 0.95, 0.01, 0.00, 0.00, 0.01, 0.00, 0.00; % BB
0.00, 0.01, 0.01, 0.01, 0.00, 0.01, 0.00, 0.00, 0.02, 0.01, 0.94, 0.00, 0.00, 0.00, 0.01, 0.00; % BE
0.00, 0.00, 0.00, 0.00, 0.01, 0.00, 0.00, 0.02, 0.00, 0.00, 0.01, 0.96, 0.00, 0.00, 0.00, 0.01; % HH
0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.01, 0.02, 0.00, 0.00, 0.00, 0.01, 0.96, 0.01, 0.01, 0.00; % BH
0.00, 0.00, 0.00, 0.00, 0.00, 0.01, 0.01, 0.02, 0.01, 0.01, 0.01, 0.00, 0.01, 0.96, 0.01, 0.00; % MV
0.00, 0.00, 0.00, 0.00, 0.00, 0.00, 0.01, 0.01, 0.00, 0.01, 0.01, 0.02, 0.01, 0.02, 0.96, 0.00; % SH
0.01, 0.01, 0.01, 0.01, 0.00, 0.00, 0.00, 0.01, 0.00, 0.00, 0.00, 0.00, 0.01, 0.00, 0.01, 0.92]; % NRW
Durch die neu Zeiteinteilung ergibt sich für den Transport Folgendes:
% Zeitschritte pro Tag
for n=1:N_t
q=1:Anzahl_Bundeslaender;
if n<N_t1
%Austauch zwischen den Bundesländern, siehe Austauschmatrix
Sneu1(n+1,q)=Sneu(n+1,q)*A;
Ineu1(n+1,q)=Ineu(n+1,q)*A;
Rneu1(n+1,q)=Rneu(n+1,q)*A;
Remneu1(n+1,q)=Remneu(n+1,q)*A;
elseif N_t1+1<=n<N_t2
Sneu1(n+1,q)=Sneu(n+1,q)*B;
Ineu1(n+1,q)=Ineu(n+1,q)*B;
Rneu1(n+1,q)=Rneu(n+1,q)*B;
Remneu1(n+1,q)=Remneu(n+1,q)*B;
elseif N_t2+1<=n<N_t3
Sneu1(n+1,q)=Sneu(n+1,q)*C;
Ineu1(n+1,q)=Ineu(n+1,q)*C;
Rneu1(n+1,q)=Rneu(n+1,q)*C;
Remneu1(n+1,q)=Remneu(n+1,q)*C;
else
Sneu1(n+1,q)=Sneu(n+1,q)*B;
Ineu1(n+1,q)=Ineu(n+1,q)*B;
Rneu1(n+1,q)=Rneu(n+1,q)*B;
Remneu1(n+1,q)=Remneu(n+1,q)*B;
endif
endfor
Ergebnis/Interpretation
[Bearbeiten]Achtung: Gegebenenfalls Plotfehler durch Octave. Das Saarland hat nicht knapp 18 Mio. Einwohner, sondern nur ungefähr 1 Mio. (siehe Bevölkerungsvektor).

Im Vergleich der Kurven des Saarlandes aus Zyklus 2 (links) und dem vorherigen Zyklus 1 (rechts), ist deutlich zu erkennen, dass die kurve der Infected viel flacher verläuft und nach dem Ende der Pandemie viel weniger Menschen die Krankheit durchlaufen haben (Susceptible noch höher). Zudem scheint in Zyklus 2 um Tag 80 herum die Zahl der Infektionen zu sinken, bevor sie noch einmal an fahrt aufnimmt und das Maximum um Tag 110 erreicht.
Um die Infektionskurve noch weiter zu senken, müsste der Austausch der Bürger zwischen dem Saarland und hauptsächlich Rheinland-Pfalz (Bild Mitte), aber auch Nordrheinwestfahlen (Bild Rechts), früher deutlich eingeschränkt werden. Betriebe könnten sofern möglich auf einen digitalen Austausch (Homeoffice/Videokonferenzen) umsteigen. Da es sich um ein diskretes Modell handelt sind auch hier Diskretisierungsfehler an Stellen zu erkennen, an denen die Steigung besonder groß ist. Es muss an dieser Stelle auch beachtet werden, dass die Susceptible des Saarlandes nicht nur in den Zustand der Infected übergehen können, sondern das Bundesland auch verlassen, bevor sie infiziert werden. Dies ist hier besonders wahrscheinlich, da im Verhältnis zu anderen Bundesländern hier weniger Menschen an Ort und Stelle verweilen und das auch noch nach einschränkenden Maßnahmen.
Optimierung
[Bearbeiten]Zyklus 3
[Bearbeiten]Ziel
[Bearbeiten]In Zyklus 3 soll gezeigt werden, welche Problematik sich aus dem Pandemieverlauf trotz Maßnahmen ergibt. Hierzu wird die Kapazität der Intensivbetten je Bundesland im Vergleich zu den Infizierten untersucht, welche eine spezielle Betreuung benötigen (Infected (intensiv)). Zudem werden diejenigen geplottet, welche positiv auf COVID-19 getestet wurden. Hier wird für die Testkapazität eine zeitabhängige Prozentzahl gewählt.
Hauptskript
[Bearbeiten]An dieser Stelle wird auch hier wieder die neue Version des Transportprozesses aufgerufen (siehe unten).
Zyklus3_wikiversity_Trans();
Da eine der wichtigsten Fragen sich damit beschäftigt, ob Pflegefälle auf Grund von COVID-19 ausreichend versorgt werden können, wird nun ein Blick auf das DIVI-Intensivregister des RKI geworfen. Die Nachfolgenden Zahlen beziehen sich auf die Gesamtzahl aktuell betreibbarer Intensivbetten und der Notfallreserve (innerhalb von 7 Tagen zusätzlich aufstellbare Intensivbetten) der Länder-Tabelle vom 09.09.2020.
%Intensivbetten pro Bundesland nach Intensivregister + Notfallzusatzbetten
for n=1:N_t
Ib(n+1,1)=0.562+0.242; %SL
Ib(n+1,2)=1.467+0.526; %RLP
Ib(n+1,3)=2.315+1.000; %HS
Ib(n+1,4)=3.333+1.499; %BW
Ib(n+1,5)=4.319+2.022; %BY
Ib(n+1,6)=1.773+0.740; %S
Ib(n+1,7)=0.917+0.383; %TH
Ib(n+1,8)=2.651+1.075; %NI
Ib(n+1,9)=1.062+0.365; %SA
Ib(n+1,10)=0.933+0.455; %BB
Ib(n+1,11)=1.275+0.455; %BE
Ib(n+1,12)=0.876+0.283;%HH
Ib(n+1,13)=0.244+0.148; %BH
Ib(n+1,14)=0.834+0.297; %MV
Ib(n+1,15)=1.047+0.286; %SH
Ib(n+1,16)=7.067+2.491; %NRW
endfor
Epidemiologischer Prozess
[Bearbeiten]Die Testkapazität wird hier je nach Zeitintervall angepasst. In diesem Fall sind mit mehr Zeit immer mehr Tests verfügbar und werden auch durchgeführt. Es wird außerdem davon ausgegangen, dass 6% an Infected auch eine Intensivbetreuung benötigen werden.
% Nicht jeder wird getestet, Dunkelziffer, Testkapazität nicht ausreichend, teilweise falsch als nicht 'testenswert' eingestuft
if n<N_t1
k=0.05;
elseif n<N_t2
k=0.12;
elseif n<N_t3
k=0.3;
else
k=0.4;
endif
% Benötigen Intensivbetten
p=0.06;
I_tested=zeros(N_t+1, Anzahl_Bundeslaender);
I_i=zeros(N_t+1, Anzahl_Bundeslaender);
Zu Beginn belaufen sich die getesteten Infizierten sowie die Intensivpatienten auf jeweils .
I_tested(1,i)=0;
I_i(1,i)=0;
Da sowohl der Test als auch eine benötigte Intensivbetreuung in der Regel (nicht immer) 14 Tage nach einer Infektion eintreten, wird der jeweilige Wert um zwei Wochen in die Zukunft verschoben.
I_tested(n+14,i)=k.*I(n,i);
I_i(n+14,i)=p.*I(n,i);
Transportprozess
[Bearbeiten]Der Transportprozess muss an dieser Stelle natürlich ebenfalls erweitert werden:
...
Ineu_tested=round(I_tested*b_0(1,i));
Ineu_i=round(I_i*b_0(1,i));
...
Ineu_i1(n+1,q)=Ineu_i(n+1,q)*A;
Ineu_tested1(n+1,q)=Ineu_tested(n+1,q)*A;
...
Ergebnis/Interpretation
[Bearbeiten]Des Weiteren ist an dieser Stelle zu beachten, dass die verfügbaren Intensivbetten (und die Reservebetten!!!) durchaus mit geplottet wurden. Allerdings ist die Anzahl der Betten im Vergleich zu den Pflegebedürftigen bereits so gering, dass der Graph nicht richtig ersichtlich ist. Die x-Achse erscheint aus diesem Grund hier blau, auch wenn die Intensivbetten nie den Wert zugeordnet bekommen. Durch Anpassung der Achse wird schließlich deutlich, welches Problem sich letztendlich für den Verlauf von COVID-19 ergibt (siehe unten).
Im Folgenden werden das Saarland und Nordrhein-Westfahlen als Beispiel für den Pandemieverlauf mit passenden (zumindest stark eindämmenden) Maßnahmen und zu schnellen Lockerungen, beziehungsweise zu späten Maßnahmen gegenübergestellt. Um Tag 65 herum sind bereits alle Intensivbetten und auch die Reservebetten belegt, die Zahl an Patienten welche eines dieser Betten benötigen steigt jedoch weiter rapide an.
Nordrhein-Westfahlen hat . Vergleicht man hier Zyklus 1 (links) mit Zyklus 3 (mittig), so ist im Verlauf kaum ein Unterschied erkennbar. Lediglich die Susceptible sind in Zyklus 3 noch weiter zurückgegangen. Ein Blick auf die Transportmatrizen und die Infektionsrate zeigen, dass sich dieses Bundesland erst sehr spät () dazu entschließt Maßnahmen zu ergreifen. Diese fallen jedoch im Vergleich zu anderen Bundesländern schwach aus. Zudem gibt es hier keine langsame Lockerung gegen Ende, sondern es wird sofort zum normalen Alltag übergegangen. Das rechte Bild zeigt deutlich, dass trotz vergleichsweise vieler Intensivbetten (vgl. Saarland) ungefähr ab Tag 62 die Kapazitäten ausgeschöpft sind.
Optimierung/Ausblick
[Bearbeiten]- Intensivbettenkapazität zeitabhängig machen
- Start der Pandemie nicht in allen Bundesländern gleichzeitig
- Tag ausgeben lassen, ab dem Intensivbettenkapazität ausgelastet ist
- Quarantäne für Infected
- Erneute Ansteckung möglich
- Offenes System: Austausch über Grenzen Deutschlands
- ...
Auch wenn das Modell immer weiter verbessert werden kann um sich der Realität weiter anzunähern, so zeigt sich doch auch hier gut der Kern des Problems. Selbst mit einschränkenden Maßnahmen ist der Verlauf der Pandemie als äußerst kritisch anzusehen. Es gibt zu wenige Intensivbetten und obwohl bestimmte Maßnahmen den Verlauf stellenweise gut gemäßigt haben, so reichen diese bei weitem nicht aus. Vor allem die Auswirkungen von Quarantänemaßnahmen wären im weiteren Verlauf der Modellierung ein interessanter Aspekt. Hierzu zählt selbst auferlegte Isolation, aber besonders auch die verordnete Quarantäne von mindestens 14 Tagen positiv getesteter Patienten.
Optimierung/Ausblick
[Bearbeiten]Das Modell lässt zwar innerhalb der Bundesländer - wenn auch je nach Beschränkungsmaßnahmen - einen Austausch der Bevölkerungsgruppen zu. Allerdings werden die Grenzen von gesamt Deutschland als geschlossen betrachtet, sodass insgesamt über die Fläche aller Bundesländer sowohl niemand dieses Gebiet verlässt als auch keiner von außerhalb einreist (egal welchen Zustands). Konkret bedeutet dies auch im unwahrscheinlichen Extremfall, dass eine sehr hohe Infektionsrate verbunden mit einer ebenso hohen Sterberate dazu führen würde, dass die Bevölkerung mit der Zeit gegen 0 geht (theoretisch), da ebenfalls keine Geburten innerhalb des Systems geschehen. Auch eine Belegung der Intensivbetten durch ausländische Infected ist somit ausgeschlossen und beeinflusst dadurch die Werte. Ebenfalls nicht einbezogen ist die Verfügbarkeit von medizinischem Personal, da in bestimmten Gebieten ein so großer Mangel herrscht, dass zwar Intensivbetten vorhanden sind, diese allerdings nicht alle betreut werden können. Um generell zu zeigen, dass beispielsweise einschränkende Maßnahmen und der vorausschauende Blick auf den Verlauf der Pandemie in anderen Bundesländern als dem eigenen hilfreich sind, ist dieses Modell angebracht. Zudem ist auch ohne zusätzliche Infected zu erkennen, dass eine Intensivbetreuung bei schwerem Verlauf große Probleme birgt.
Aufgaben Vorlesung
[Bearbeiten]Ziel
[Bearbeiten]Kontaktmodell
[Bearbeiten]Ziel
[Bearbeiten]Im Folgenden wird die räumliche Ausbreitung einer Infektion ausgehend von einem Infizierten (Indexpatienten) durch ein Kontaktmodell dargestellt. Die Personen bewegen sich ausgehend von einer festgelegten Startposition innerhalb eines generierten Gitters mit individueller Geschwindigkeit und Zielrichtung in einem festgelegten zeitlichen Rahmen durch den Raum. Durch Kontakt mit Infizierten innerhalb eines festgelegten Radius soll automatisch eine Infizierung erfolgen sodass abgesehen werden kann, wie schnell sich die Infektion unter dieser eingegrenzten Gruppe ausbreitet.
Skript
[Bearbeiten]Zunächst werden hierfür die wichtigsten Parameter festgelegt. Es wird ein Gitter erstellt (hier 10x20 Punkte), auf welchem gesunde Menschen/Pünktchen zunächst noch streng geordnet platziert sind. Es wird außerdem festgelegt, dass beispielweise zwei große Zeitschritte durchlaufen werden, welche jeweils noch einmal in zwei kleinere Zeiteinheiten unterteilt werden. Wie nah sich eine gesunde Person zu einer infizierten befinden muss um sich ebenfalls anzustecken, wird durch den Infektionsradius festgelegt. Wird dieser Abstand unterschritten, so erfolgt nach späterer Definition eine sichere Ansteckung. Damit dies geschehen kann, wird zunächst eine Person als infiziert festgelegt, welche sich unter den gesunden im Gitter befindet.
%Anzahl von Punkten in x und y Richtung
Nx=10;
Ny=20;
Zeitschritte=2; %pro Zeiteinheit T=1
radiusInf=1.3 %Infektionsradius, wird dieser unterschritten, so steckt sich das nächste Kästchen an
T=2 ; %Die Berechnung wird für so viele Zeiteinheiten laufen
g=0.5; %Geschwindigkeit Fußgänger/Kästchen -> Geschwindigkeit x Zeitschritte = Wie weit kommt mein Punkt voran? z.B. 0,5 in einem Zeitschritt
x=[1:Nx];
y=[1:Ny];
dt=1/Zeitschritte;
[x,y]=meshgrid (x,y) ; %Gitter wird erstellt
%Position erster Infizierter
x2=9;
y1=3;
xInf=x(1,x2);
yInf=y(y1,1);
IndInf2=x2 %Index
IndInf1=y1;
IndInf1neu=IndInf1 ;
IndInf2neu=IndInf2 ;
close all;
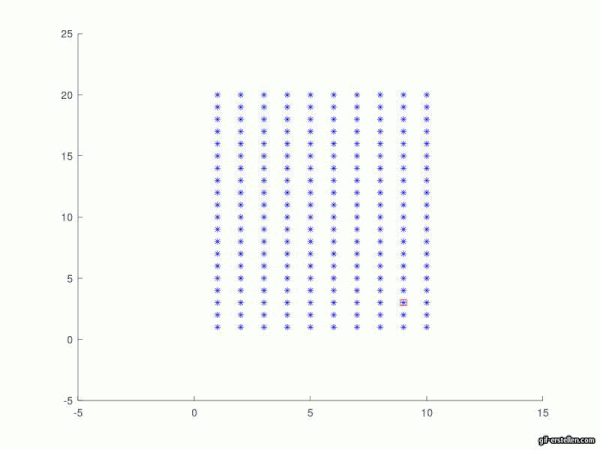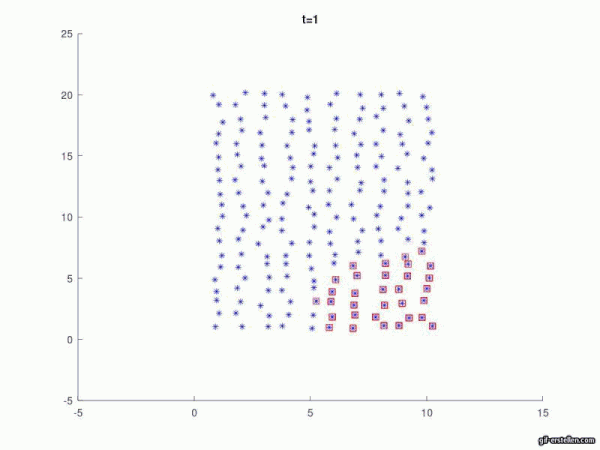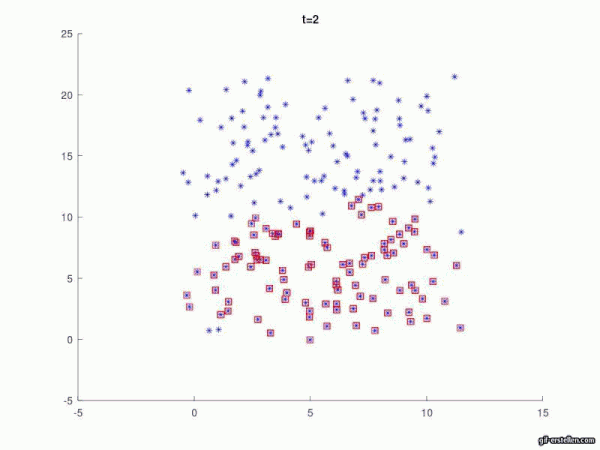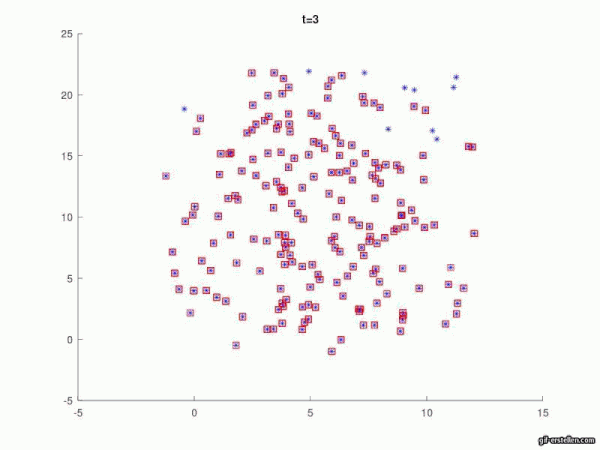
Hier wird Bild 1 geplottet. Dies entspricht dem Ausgangszustand und enthält daher den Indexpatienten (Patient 0). Optional kann das Bild automatisch abgespeichert werden.
figure (1)
hold on;
plot(x, y, '*b') ;
plot(IndInf2, IndInf1, 'sr');
axis([-5 15 -5 25]); % Achsenskalierung wird festgelegt
%mit folgenden Befehlen kann man Bilder speichern und für weitere Bearbeitung verwenden (Videos, Animationen):
%test=["Fig_", num2str(1), ".jpg"]
%saveas(1, test)
hold off;

Um die Ausbreitung zu zeigen, muss eine Zeitschleife erstellt werden, welche alle Zeiteinheiten durchläuft und mehrere Befehle abdeckt:
- Ab der Hälfte der Zeitschritte sollen sich die Personen in Param-facher (hier fünffacher) Geschwindigkeit bewegen
- Alle Personen bewegen sich in unterschiedliche, zufällige Richtungen in ebenfalls zufälliger Geschwindigkeit
- Dies soll sich nach jeder Zeiteinheit erneut ändern
- Wir der Infektionsradius von einer gesunden Person zu einer infizierten unterschritten, so steckt sich diese ebenfalls an
%Zeitschleife für Zeitschritte (Anzahl festgelegt, s.o.) pro eine Zeiteinheit T:
Param=1
for i=1:T*Zeitschritte
if i==floor(T*Zeitschritte/2) %== prüft
Param=5;
endif
neuPosX=g.*rand(Ny,Nx)-0.5*g; % so soll sich das x verhalten, random sorgt für zufällige Bewegung rand(Ny,Nx)-0.5*g = Zufallszahl zw. -0.5g u. 0.5g
neuPosY=g.*rand(Ny,Nx)-0.5*g;
neuX=x.+Param*(neuPosX)*i*dt;
neuY=y.+Param*(neuPosY)*i*dt;
Um zu sehen wie schnell und in welche Richtung sich die Pünktchen jeweils bewegen, kann der Befehl quiver genutzt werden. Bei den anderen Plots muss darauf geachtet werden, dass figure 1 und 2 bereits belegt sind.
%Mit diesem Befehl kann man sich die Geschwindigkeitspfeile anschauen
figure (2)
quiver (neuX.-x,neuY.-y);
hold off
figure (i+2) %hier i+2, da figure 1 und 2 bereits belegt sind
title (['t=' num2str(i*dt)]) % num2str(A) konvertiert numerische Ordnung in Charakteristische, um Zahlen zu ordnen. Hilfreich um Plots mit numerischen Werten zu betiteln.
axis([-5 15 -5 25]);
hold on
plot(neuX,neuY, '*b') ;
%Speichern der Bilder: optional
%test=["Kontaktmodell_kommentiert_", num2str(i),".jpg"]
%saveas(i, test)
Im nächsten Schritt muss der wichtigste Teil des Programms festgelegt werden. Jedes Pünktchen, dass sich durch den Raum bewegt, muss pro kleinschrittiger Zeiteinheit auf seinen Abstand zu allen anderen Pünktchen geprüft werden, um eine Verbreitung der Krankheit modellieren zu können. Dies bedeutet, dass der Infektionsradius für jedes Pünktchen individuell gilt (aber für alle gleich). Treffen sich in diesem Radius also zwei gesunde Pünktchen, so soll natürlich keine Infektion erfolgen, da keiner der beiden Krankheitsträger ist. Bei einem gesunden Pünktchen, welches innerhalb dieses Radius jedoch einem infizierten begegnet soll immer eine Infizierung stattfinden. Alle neu Infizierten sollen dann eine Markierung wie das Indexpatientpünktchen (rotes Kästchen) erhalten. Gleichzeitig muss darauf geachtet werden, dass der Kontakt zweier bereits infizierter Pünktchen nicht dazu führt, dass diese sich im Modell immer "mehr infizieren" (d.h. sich überlappende Markierungen ausschließen), da der Infektionszustand in sich nicht abgestuft ist und man sich nicht mehr als infizieren kann.
%neue Infizierungen:
%In Vektoren IndInf1, IndInf2 sind die i- und j-Indexe der (x,y)-Koordinaten der bereits Infizierten gespeichert
zahler=1;
for k1=1:length(IndInf1) % Länge des aktuell gespeicherten Infiziertenindex, wird immer weiter ergänzt, k1=Index 1 bis Ende
for j=1:Nx
for l=1:Ny
%die aktuellen Positionen der Fußgänger-Punkte werden mit den Positionen der Infizierten abgeglichen (Abstand wird berechnet)
%norm drückt abstände aus, abstand von jeder einzelnen person im koordinatensystem wird mit infizierter Person verglichen
abstand=norm ( [neuX(l,j)-neuX(IndInf1(k1),IndInf2(k1)), neuY(l,j)-neuY(IndInf1(k1),IndInf2(k1))]); %eckige Klammer = Vektor
%wenn der Abstand zum Infizierten unter dem Ansteckunsradius liegt, wird der l,j-te Punkt infiziert (mit rotem Quadrat gekennzeichnet)
if abstand<radiusInf
abstand; %Rückgabewert
plot(neuX(l,j),neuY(l,j), 'sr') ;
%---------------------------
k2=1;
while k2 <=length(IndInf1)&&(l!=IndInf1neu(k2) || j!=IndInf2neu(k2))
k2=k2+1;
endwhile
if k2 >length(IndInf1) %wenn k2 nicht in Vektor der schon Infizierten liegt (durch alle Indexe gegangen und nicht gefunden) dann:
IndInf1neu=[IndInf1neu l]; %Indexe werden einfügt in Vektor
IndInf2neu=[IndInf2neu j];
endif
endif
endfor
endfor
endfor
IndInf1=IndInf1neu;
IndInf2=IndInf2neu;
hold off
lengt=length(IndInf1);
endfor
%Zeitschleife abgeschlossen
Plots
[Bearbeiten]In den folgenden drei Animationen können veränderte Ausbreitungen der Krankheit auf Grund einer Variation des Infektionsradius beobachtet werden. Links wurde der Wert auf 0.7 gesetzt, mittig auf 1.0 und rechts auf 1.3. Alle anderen Werte blieben unverändert.
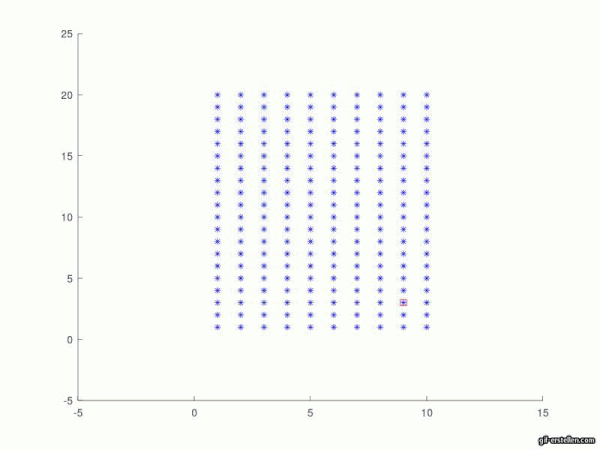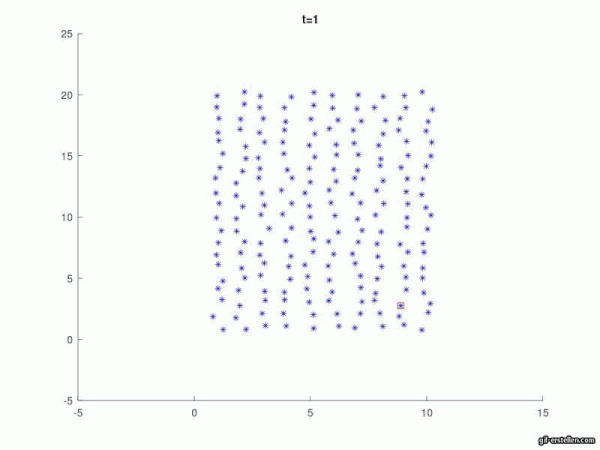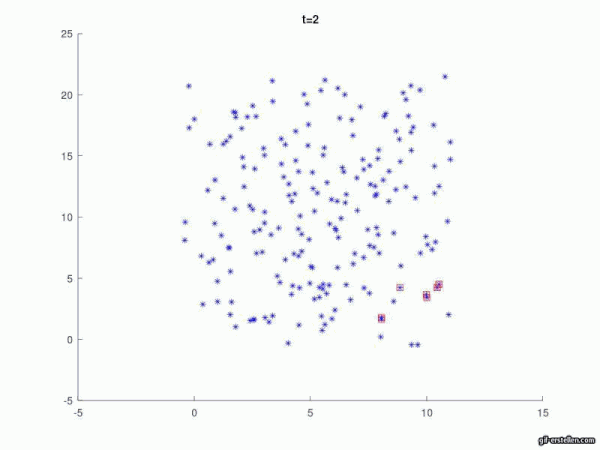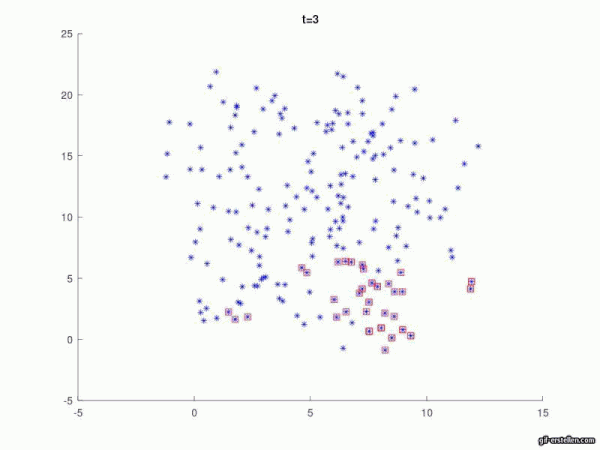

Wie zu erwarten führt ein größerer Infektionsradius zu einer schnelleren Ausbreitung der Krankheit. Hieraus lässt sich zum Beispiel ableiten, dass es wichtig ist die Abstandsregeln einzuhalten und zu wissen, wie groß der Infektionsradius ungefähr ist.
In den nächsten drei Animationen kann beobachtet werden, welchen Einfluss die Position des Indexpatienten bei gleichbleiendem Infektionsradius auf die Infektionsausbreitung hat.
Es ist erkennbar, dass ein Indexpatient mit einer höheren Anzahl ihn umgebender Anfälliger (Susceptibles) die Krankheit schneller weitergeben kann. Am Rand oder den Ecken des Gitters platziert ist es ihm hingegen möglich den anderen Pünktchen aus dem Weg zu gehen, wodurch sich die Ausbreitung verlangsamt. Aus dieser Animation kann geschlussfolgert werden, dass eine Infektion leichter vermieden werden kann, wenn man sich nicht mitten in einer Menschenmenge befindet.
Optimierung
[Bearbeiten]In diesem Modell wird davon ausgegangen, dass eine Infizierung in Folge einer Unterschreitung des festgelegten Radius sicher ist. Bezogen auf COVID-19 muss jedoch davon ausgegangen werden, dass ein Kontakt zwischen Menschen unter Einhaltung der Hygieneregeln (Masken, Lüften, etc.) ein geringeres Infektionsrisiko birgt, als ein Miteinander ohne Beachtung dieser Maßnahmen. Mit Hilfe der Wahrscheinlichkeit könnte festgelegt werden, in wie vielen Fällen die Infektion der Kontaktperson tatsächlich erfolgt (beziehungsweise nicht). Des Weiteren ist es nicht besonders realitätsnah, dass alle Personen in jedem kleinteiligen Zeitschritt ihre Laufrichtung random (bzw. überhaupt) ändern, da Menschen meist zielgerichtet und individuell gehen. Zudem ist zu beachten, dass keine Pünktchen das System verlassen, oder neu hinzugefügt werden. Es handelt sich somit um ein geschlossenes System ohne Tod (Removed) und Geburt, indem allerdings auch die Genesung nicht berücksichtigt wird. Alle im System enthaltenen Pünktchen befinden sich somit entweder im Zustand vor der Infektion (Susceptible) oder sind und bleiben infiziert (Infected).
Räumliche Diffusion
[Bearbeiten]Den folgenden Modellierungen ist die räumliche Diffusion zu Grunde gelegt. Dies meint die zufällige Bewegung von Teilchen, sodass ein vorliegender Konzentrationsunterschied ausgeglichen wird und mit der Zeit eine vollständige Durchmischung (Gleichgewicht) erreicht wird. Dieser Prozess wird mathematisch durch
beschrieben, wobei und wie folgt definiert sind:
die unbekannte Teilchenkonzentrationsdichte/Temperaturdichte (räumliche und zeitliche Variable)
der ortsabhängige Diffusion-/Wärmeleitkoeffizient, dieser beschreibt die Geschwindigkeit der Ausbreitung des Stoffes
Laplace
[Bearbeiten]Die Laplace-Gleichung ist eine der einfachsten partiellen Differentialgleichungen. Es ist möglich die Lösung als Funktion anzugeben. Die sogennante Fundamentallösung der Laplace Gleichung lautet
Stationäre Lösung
[Bearbeiten]Zunächst wird nur der stationäre Fall betrachtet. Dies bedeutet, dass unabhängig von der Zeit ist und somit nur von den räumlichen Variablen abhängt (). Gilt zudem , so folgt daraus die Laplace-Gleichung .
Skript
[Bearbeiten]Zunächst wird ein Gitter festgelegt. Dieses umgeht hier die Stelle , da die Fundamentallösung singulär im Ursprung des Koordinatengitters ist. Das heißt für den Punkt , beziehungsweise in mehreren Dimensionen, erreicht die Fundamentallösung den Wert . Wenn die Schrittweite allerdings klein genug gewählt wird und der Bereich um den Ursprung des Koordinatensystems ebenfalls eng genug gefasst wird, so ist das fehlende Gebiet im Plot nicht weiter störend.
step=0.05; %Schrittgröße
X=[-2:step:-0.01, 0.01:step:2]; %Gebietseingrenzung um Ursprung des Koordinatensystems
Y=[-2:step:-0.01, 0.01:step:2];
[x,y]=meshgrid(X,Y); %verbindet x und y in Matrix, so dass Punktegitter entsteht
Für den zweidimensionalen Raum handelt es sich bei der Lösung um eine logarithmische Funktion. Die Formel der Fundamentallösung für lautet :
phi=@(x,y) -1/(2*pi)*log(sqrt(x.^2+y.^2));
Nun muss die Lösung noch geplottet werden. Eine Speicherung des Bildes ist optional.
figure (1)
meshc(x,y,phi(x,y))%3D Plot
grid on
colormap winter; %farblich blau gelb grün
title('Fundamentallösung Laplace')
xlabel('x')
ylabel('y')
zlabel('z')
axis([-2 2 -2 2 -0.2 1]) %feste Achen für Vergleich besser
%Speichern der Bilder
test=["Fundamentallösung Laplace.jpg"]
saveas(1, test)
Plot
[Bearbeiten]
Instationäre Lösung
[Bearbeiten]Nun wird die instationäre Lösung der Laplace-Gleichung modelliert. Diese unterscheidet sich von der Stationären durch den Einbezug der Zeit .
Skript
[Bearbeiten]Zunächst wird wie zuvor ein Gitter erstellt, welches den Ursprung des Koordinatensystems auf Grund der Singularität umreißt, dies entspricht noch der vorherigen Vorgehensweise.
step=0.05; %Schrittgröße
X=[-2:step:-0.01, 0.01:step:2]; %Gebietseingrenzung um Ursprung des Koordinatensystems
Y=[-2:step:-0.01, 0.01:step:2];
[x,y]=meshgrid(X,Y); %verbindet x und y in Matrix, so dass Punktegitter entsteht
Im nächsten Schritt wird zunächst ein fester Diffusionskoeffizient a festgelegt () und es wird ebenfalls eine Zeit eingegrenzt, in welcher die Diffusion betrachtet wird. In diesem Fall handelt es sich um vier größere Zeiteinheiten, welche noch einmal in vier weitere Untereinheiten eingeteilt sind. Dies liefert dann später 16 Bilder.
a=0.5; % Geschwindigkeit der Diffusion
T=4;
Schritte_T=4
dt=1/Schritte_T;
t_0=0.25
Mit Hilfe einer Zeitschleife wird die Lösung für jeden kleinen Zeitschritt berechnet, geplottet und als Bild gespeichert. Da die Änderung der Teilchenkonzentration so besser zu erkennen ist, wurde hier an Stelle der colormap winter auf die colormap spring zurückgegriffen, da der Kontrast zwischen gelblich-orange und pink größer ist.
for m=0:T*Schritte_T
t=t_0+m*dt
psi=@(x,y,t) 1/(4*pi*a*t)*exp(-sqrt(x.^2+y.^2)/(4*a*t));
figure (m+1)
meshc(x,y,psi(x,y,t))%3D Plot
grid on
colormap spring; %farblich pink orange/gelb
title(['Laplace in t=',num2str(t_0+m*dt)])
xlabel('x')
ylabel('y')
zlabel('z')
axis([-2 2 -2 2 0 1.1]) %feste Achen für Vergleich besser
%Speichern der Bilder
test=["Instationäre Fundamentallösung Laplace_", num2str(m+1),".jpg"]
saveas(m+1, test)
endfor
Plot
[Bearbeiten]Auch wenn der große "Hügel", also der Ballungsraum der Teilchen zu Beginn, relativ schnell nach außen diffundiert und bereits in kaum mehr zu erkennen ist, so ist die Wahl der gesamten Zeitspanne bis gut dazu geeignet um zu zeigen, dass die Lösung mit der Zeit gegen geht:

Poisson
[Bearbeiten]Ziel
[Bearbeiten]Stationärer Fall
[Bearbeiten]Wird jedoch nur der stationäre Fall , also der Gleichgewichtszustand betrachtet, so ist die Zeitableitung gleich Null. Für die folgende Modellierung wird ein rechteckiges Gebiet betrachtet.
Mit Hilfe der Fundamentallösung der Laplace-Gleichung kann man auch Lösungen der Poisson-Gleichung konstruieren, da die Poisson-Gleichung die inhomogenen Laplace-Gleichung ist (). Dies geschieht durch die Faltung der Fundamentallösung wird mit der Funktion der rechten Seite . Als Voraussetzung muss gelten, dass einen kompakten (beschränkten)Träger inerhalb des Gebietes besitzt.
Skript
[Bearbeiten]Im folgenden wird zuerst ein rechteckiges Gebiet mit Hilfe eines Gitters (Befehl meshgrid(X,Y)) definiert, wobei die Schrittweite mit festgelegt ist.
h=0.1; %Schrittgröße
X=[0:step:10]; %von 0 bis 10 in 0.1er Schritten
Y=[0:step:10];
[x,y]=meshgrid(X,Y); %verbindet x und y in Matrix, so dass Punktegitter entsteht
Die folgende Funktion der rechten Seite (hier Anfangsfunktion1(x,y)) gibt eine Quelle an, wonach an dieser festgelegten Stelle (oder an mehreren) eine höhere Konzentration an Teilchen herrscht:
function wert=Anfangsfunktion1(x,y)
if sqrt((x.-8).^2+(y.-8).^2)<=1.10 wert=1;
elseif sqrt((x.-2).^2+(y.-2).^2)<=1.00 wert=1;
else wert=0;
endif
endfunction
Zuerst werden die Werte der Fundamentalfunktion für jeden Gitterpunkt um ein festes verschoben und in der Matrix gespeichert. Dazu wird als Matrix mit konstanten Einträgen durch Konstruktion einer Schleife definiert, damit die Differenz der Funktionsargumente als Differenz von Matrizen möglich ist.
%Die Funktionswerte müssen dann in jdem Gitterpunkt f(x(j,i),y(j,i)) in eine Matrix gespeichert werden:
for i=1:(length(X))
for j=1:(length(Y))
f(j,i)=1*Anfangsfunktion1(x(j,i),y(j,i));
endfor
endfor
%Jetzt: feste Marizen x* und y* mit konstanten Einträgen festlegen
for i=1:(length(X))
for j=1:(length(Y))
xstar=x(j,i)*ones(length(Y),length(X));
ystar=y(j,i)*ones(length(Y),length(X));
Wichtig: Wenn setzen wir für , da sonst singulär ist (d.h. ). Das Herausschneiden dieses einzigen Punktes im stetigen Fall spielt im integral keine große Rolle, da diese Menge in Betrachtung von das Maß 0 hat. Das doppelte Integral wird mit der Trapezregel berechnet.
%Werte der Fundamentalfkt. werden um festes x*, y* verschoben
%Abspeicherung in Matrix phi
%Phi mit verschobenem Argument:
phi=-log(sqrt((xstar.-x).^2+(ystar.-y).^2))/(2*pi);
phi(j,i)=0;
%numerische Integration
I(j,i) = trapz(Y,trapz(X,phi.*f,2));
endfor
endfor
Im Folgenden werden dann die Lösung der Poissongleichung sowie die Funktion der rechten Seite geplottet:
%%-------- Plot 1 ----------------------
figure (1)
meshc(x,y,I(:,:,1)) %3D Plot
grid on
colormap winter; %farblich blau gelb grün
title('Lösung der Poissongleichung')
xlabel('x')
ylabel('y')
zlabel('z')
%Speichern der Bilder
test=["Lösung der stationären Poissongleichung.jpg"]
saveas(1, test)
%%-------- Plot 2 ----------------------
figure (2)
surfc(x,y,f,'FaceColor','b')
title ('Funktion der rechten Seite')
xlabel('x')
ylabel('y')
%Speichern der Bilder
test2=["Funktion der rechten Seite.jpg"]
saveas(2, test2)
Plot
[Bearbeiten]Instationäre Diffusion mit Anfangswertproblem
[Bearbeiten]Mit einem konstanten Diffusionskoeffizienten folgt die homogene instationäre Differenzialgleichung , für welche die Lösungsformel existiert, wobei das Quadrat der euklidischen Norm von ist.
Wie bei der Laplace-Gleichung kann man mit Hilfe der Fundamentallösung die Integrallösungsformel für das sogenannte Anfangswertproblem der Diffusionsgleichung definieren:
Die Funktion beschreibt somit den anfänglichen Zustand der Lösung für den Zeitpunkt . Die Lösung für ist widerum durch die Faltung der Fundamentallösung mit der Anfangsfunktion gegeben:
Skript
[Bearbeiten]h=0.1; %Schrittgröße
X=[0:step:10]; %von 0 bis 10 in 0.1er Schritten
Y=[0:step:10];
[x,y]=meshgrid(X,Y); %verbindet x und y in Matrix, so dass Punktegitter entsteht
a=0.3; % Geschwindigkeit der Diffusion
T=2;
Schritte_T=3
dt=1/Schritte_T;
t_0=1
Zunächst muss eine neue Anfangsfunktion mit kompaktem Träger aufgestellt und die Funktionswerte von müssen erneut in jedem Gitterpunkt der Matrix gespeichert werden.
function wert=Anfangsfunktion(x,y)
if sqrt((x.-2).^2+(y.-2).^2)<=0.15 wert=1;
elseif sqrt((x.-8).^2+(y.-6).^2)<=0.26 wert=1;
elseif sqrt((x.-1).^2+(y.-5).^2)<=0.15 wert=1;
elseif sqrt((x.-6).^2+(y.-1).^2)<=0.09 wert=1;
else wert=0;
endif
endfunction
%Die Funktionswerte müssen dann in jedem Gitterpunkt f(x(j,i),y(j,i)) in eine Matrix gespeichert werden:
for i=1:(length(X))
for j=1:(length(Y))
u0(j,i)=1*Anfangsfunktion(x(j,i),y(j,i));
endfor
endfor
Die Berechnung der Fundamentallösungsmatrizen zu den verschiedenen Zeitpunkten erfolgt durch eine Zeitschleife. Die Werte der Fundamentalfunktion werden für jeden Gitterpunkt um ein festes verschoben und in der Matrix gespeichert.
Da die Zeit mit betrachtet wird herrscht hier keine Singularität (sonst ), da die Funktion auch für y=x in diesem Punkt beschränkt ist. Das doppelte Integral wird mit der Trapezregel berechnet.
%Jetzt: feste Marizen x* und y* mit konstanten Einträgen festlegen
for m=0:T*Schritte_T
t=t_0+m*dt
for i=1:(length(X))
for j=1:(length(Y))
xstar=x(j,i)*ones(length(Y),length(X));
ystar=y(j,i)*ones(length(Y),length(X));
%Werte der Fundamentalfkt. werden um festes x*, y* verschoben
%Abspeicherung in Matrix psi
%Psi mit verschobenem Argument:
psi=(1/(4*pi*a*t))*exp(-(((xstar.-x).^2+(ystar.-y).^2))/(4*a*t));
%numerische Integration
I(j,i,m+1) = trapz(Y,trapz(X,psi.*u0,2));
endfor
endfor
Die einzelnen zeitabhängigen Zustände der Diffusion werden mit Hilfe einer Schleife geplottet und je nach Bedarf abgespeichert. Hier ist zu beachten, dass der Titel je nach konstantem Diffusionskoeffizienten a angepasst werden muss.
%%-------- Plot 1 ----------------------
figure (m+1)
meshc(x,y,I(:,:,m+1)) %3D Plot
grid on
colormap winter; %farblich blau gelb grün
title(['Für a=0.1 Lösung in t=', num2str(t_0+m*dt)])
xlabel('x')
ylabel('y')
zlabel('z')
axis([0 10 0 10 0 0.16]) %feste Achsen für Vergleich besser
%Speichern der Bilder
%a03=["Fig_", num2str(m+1),".jpg"]
%saveas(m+1, a03)
endfor
Zusätzlich soll der folgende Plot die Anfangsfunktion ausgeben:
%%-------- Plot 2 ----------------------
figure (33)
surfc(x,y,u0,'FaceColor','b')
title ('Funktion der rechten Seite')
xlabel('x')
ylabel('y')
axis([0 10 0 10 0 1.2])
Plots
[Bearbeiten]Der zweite Plot stellt die Funktion der rechten Seite dar:

Im Folgenden wurde durch Veränderung von a die Geschwindigkeit der Diffusion beeinflusst:
Optimierung
[Bearbeiten]Das Modell ist gut dazu geeignet um zu zeigen, dass ein Hotspot an Infizierten auf seine Umgebung umgreift und sich die Anzahl an Infizierten vom Zentrum dieses Ballungsgebiet nach außen hin verbreitet. In Bezug auf COVID-19 ist dieses Modell allerdings so noch unpassend, da keine neuen Infektionsherde entstehen. Zudem ist es nicht direkt möglich ein Gleichgewicht zu erreichen, da sich die Zustände (Susceptible, Infected, Removed) eigentlich ständig und nicht für alle gleich ändern. Des Weiteren kommt hinzu, dass die Massenverbreitung nicht in alle Richtungen gleichmäßig verläuft.
Wachstumsmodelle
[Bearbeiten]Logistisches Wachstumsmodell
[Bearbeiten]Das logistische Wachstumsmodell beschreibt, dass der Anstieg an Infektionen im neuen Zeitpunkt proportional zum Bestand und der freien Kapazität ist. Dabei ist die logistische Wachstumsrate (Infektionsrate). Dies bedeutet also, dass eine große freie Kapazität () in Verbindung mit wenigen Infizierten () einen ebenso geringen Anstieg beschreibt wie die Kombination aus vielen Infizierten und wenig freier Kapazität. Formal ausgedrückt sieht dies wie folgt aus:
Über die Division von und den Grenzübergang erhält man die Differenzialgleichung . Zudem gilt die Anfangsbedingung , da es zu Beginn bereits Infizierte gibt und diese Anzahl festgelegt werden muss. Es ergibt sich die logistische Funktion als Lösung.
Das logistische Wachstumsmodell, auch SI-Modell genannt (siehe unten), eignet sich aus folgenden Gründen gut als Modell für die Abbildung von COVID-19:
- Die Anzahl an Infizierten ist hier auf die Gesamtbevölkerung () beschränkt. Das bedeutet, dass die Infizierten in ihrer Anzahl nicht über die gesamte Population im System hinauswachsen kann (). Genauer verweist dies auf ein Maximum. Die Infektion stoppt, wenn eine Sättigung eingetreten ist () und niemand mehr neu Infiziert werden kann. Dies ist vor allem der große Vorteil in Bezug auf das Exponentielle Wachstumsmodell, in welchem der Anstieg lediglich proportional zum Bestand ist. Dies führt dazu, dass es keine obere Grenze an Infektionen gibt.
- Die Population der Infizierten wächst langsamer, je weniger freie Kapazität () im System herrscht. Das bedeutet, dass mit fortschreitender Zeit die Zahl der Neuinfektionen sinkt.
Folgende Punkte werden in diesem Modell außer Acht gelassen:
- Geburten im System: Die Gesamtpopulation bleibt konstant.
- Tod durch andere Ursachen: Tote welche sich nie infiziert haben werden ebenfalls nicht mit einbezogen.
Kompartimentmodelle
[Bearbeiten]Kompartimentmodelle eignen sich zur Beschreibung der Infektionsverbreitung. Sie teilen die Gesamtpopulation hierbei in zwei (oder mehrere) Gruppen ein. Zunächst wird das SI-Modell, bei welchem es sich um das einfachste Kompartimentmodell handelt, näher betrachtet.
SI-Modell
[Bearbeiten]Das SI-Modell teilt die Gesamtpopulation in zwei Gruppen ein und zwar in Susceptible (Infektionsanfällige) und Infected (kummulierte Infizierte). Die Bevölkerung besteht somit aus Menschen, welche das Virus noch nicht in sich getragen haben und solchen, die die Infektion haben oder hatten. Zwischen aktuell infiziert, genesen und tot wird hier kein Unterschied gemacht. S und I haben also einen besonderen Zusammenhang. Dabei ist die Infektionsrate und die Gesamtbevölkerung (obere Grenze der freien Kapazität). Es gilt:
Zum Startzeitpunkt gilt zudem für S und I, dass es einen Indexpatienten oder wie im Folgenden eine Indexgruppe gibt. Das heißt die Susceptibles entsprechen nicht der maximalen freien Kapazität (Gesamtbevölkerung) und die Infected haben bereits eine geringe Anzahl erreicht. Es gilt somit:
Nach dem Erhaltungsgesetz folgt zudem aus , dass die Gesamtpopulation konstant erhalten bleibt. Es können sich nicht mehr Menschen infizieren, als im System vorhanden sind und dadurch wird die obere Grenze der freien Kapazität () nie überschritten.
Skript
[Bearbeiten]Zunächst wird der zeitliche Rahmen festgelegt, in welchem der Ansteckungsverlauf, beziehungsweise die Wechselwirkung zwischen den Susceptibles und den Infected beobachtet werden soll.
t_0=0; %Startzeitpunkt
T=150; %Zeitliche Eingrenzung
t=(t_0:0.1:T); %Zeitspanne
Anschließend muss die Bevölkerung - also die Gesamtkapazität aller, die sich infizieren können - festgelegt werden. Im Zeitpunkt (Start) wird die Anzahl an Indexpatienten festgesetzt, von welchen aus sich das Virus mit fortschreitender Zeit bis zum Zeitpunkt verbreitet. Die Infektionsrate ist durch ein festes gegeben.
B=55400; %Bevölkerung/Obere Kapazitätsgrenze
I_0=16/1000; %Anfangsinfizierte, I(t_0)
k=0.3219; %Infektionsrate
Nun wird ein Spaltenvektor mit den Startwerten erzeugt. Dieser enthält somit die Susceptibles (ohne die Indexpatienten) und die Infected (nur die Indexpatienten).
y_0=[B-I_0;I_0]; %Spaltenvektor der Startwerte
Nun wird die Funktion f der rechten Seite des DGL-Systems definiert und anschließend die Lösung generiert.
% Funktion f
f=@(y,x) [-k*y(1)*y(2)/B;k*y(1)*y(2)/B;]; %S sinken, I steigen
%numerische Lösung
y=lsode(f, y_0, t);
Im letzten Schritt werden die Graphen geplottet. Ein Speichern der Bilder ist optional.
%Plot
figure(1)
plot(t,y(:,1),t,y(:,2))
hold on
title(['Infektionsrate k=', num2str(k)])
xlabel('Zeit t')
axis([t_0 T 0 B+5000])
ylabel('Bevölkerung (in Tausend)')
legend(['Susceptible';'Infected (kummuliert)'],"location","east")
hold off
%Speichern der Bilder (optional):
test=["SI-Modell Infektionsrate k=", num2str(k),".jpg"]
saveas(1, test)
Plot
[Bearbeiten]SIR-Modell
[Bearbeiten]Rate wechseln. Das SIR-Modell enthält neben den Susceptible und den Infected zusätzlich noch die Gruppe der Removed (R). In diesem Fall gilt:
ist hier die Wechselrate zu den Genesenen, da der Umkehrwert der Genesungszeit .
Es gilt hier das Erhaltungsgesetz , das beschreibt, dass die Gesamtpopulation konstant erhalten bleibt.
Skript
[Bearbeiten]Nachdem der zeitliche Rahmen festgesetzt wurde (wie im SI-Modell), müssen die Anfangswerte ergänzt werden. Es wird also festgelegt, dass es zu Beginn im Zeitpunkt noch keine Removed gibt.
B=55400; %Bevölkerung/Obere Kapazitätsgrenze
I_0=16/1000; %Anfangsinfizierte, I(t_0)
R_0=0; %Genesene + Tote zu Beginn
Zusätzlich zur Wechselrate wird hier die Genesungsrate festgelegt. Dies dient vor allem dem Plot am Ende, da die Titel der Bilder sonst die Wechselrate als Dezimalzahl enthalten und nicht als Bruch angegeben werden. Um ablesen zu können wann jedoch der Zustand der Infected zu den Removed übergeht, ist eine Angabe als Bruch jedoch vorteilhafter.
k=0.3219; %Infektionsrate
g=15; %Genesungsrate
w=1/g; %Wechselrate zu den Removed / hier Infektion nach 14 Tagen überwunden
Der Spaltenvektor muss an dieser Stelle um die Startwerte der Removed ergänzt werden.
y_0=[B-I_0;I_0;R_0]; %Spaltenvektor der Startwerte
Die nächsten Schritte laufen analog zum SI-Modell ab.
% Funktion f
f=@(y,x) [-k*y(1)*y(2)/B;k*y(1)*y(2)/B-w*y(2);w*y(2)]; %S sinken, I haben Maximum, R steigen
%numerische Lösung
y=lsode(f, y_0, t);
Aus den bereits genannten Gründen muss beim Plotten darauf geachtet werden, dass sich der Befehlnum2str im Titel auf die Genesungsrate bezieht.
%Plot
figure(1)
plot(t,y(:,1),t,y(:,2),t,y(:,3))
hold on
title(['Wechselrate w=1/', num2str(g)])
xlabel('Zeit t')
axis([t_0 T 0 B+5000])
ylabel('Bevölkerung (in Tausend)')
legend(['Susceptible';'Infected (aktuell)';'Removed'],"location","east")
hold off
%Speichern der Bilder (optional):
test=["SIR-Modell Wechselrate w=", num2str(w),".jpg"]
saveas(1, test)
Plot
[Bearbeiten]In diesen Plots können die Auswirkungen der Änderung von (Infektionsrate) bei gleichbleibender Wechselrate beobachtet werden. Es wird deutlich (besonders in der Animation rechts), dass bereits kleinste Änderungen einen großen Effekt auf den Verlauf der Pandemie haben. Bezogen auf COVID-19 bedeutet dies, dass jede noch so kleine Chance die Infektionsrate niedrig zu halten genutzt werden muss und wirklich jede Handlung in Bezug darauf einen merklichen Effekt hat. Je höher ist, desto größer ist die Welle an Infizierten und desto früher und steiler kommt der Anstieg bezogen auf die laufende Zeit .
Im Folgenden bleibt (Infektionsrate) konstant, jedoch ändert sich die Wechselrate . Es zeigt sich, dass bereits ein Unterschied von zwei Tagen zu viel mehr Infektionen führt. Bezogen auf COVID-19 können wir auf die Dauer der Infektion zwar keinen Einfluss nehmen, jedoch zeigt sich hier, dass die Quarantäne eine wichtige Rolle spielt und es maßgeblich ist, sich im Falle einer Infektion an diese Einschränkung zu halten.
Verbindung zum SI-Modell
[Bearbeiten]Im Folgenden sieht man den Zusammenhang zwischen dem SI- und dem SIR-Modell. Dem SIR-Modell wurden hierfür die kummulierten Infizierten hinzugefügt:

Optimierung
[Bearbeiten]Das SI-Modell sowie das SIR-Modell beziehen keine Geburten sowie Todesfälle außerhalb einer COVID-19 Infizierung mit ein. Zudem werden hier noch keine beschlossenen (Hygiene)maßnahmen beachtet, welche die Infektionsrate zeitabhängig beeinflussen. Auch die Wechselrate zu den Genesenen ist nicht für alle Menschen gleich. Es gibt durchaus Fälle, in denen eine Infektion viel länger anhält als durchschnittlich bei anderen. Des Weiteren würden nach diesem Modell auch Infektionen von Menschen weitergegeben werden, welche alleine zu Hause in Quarantäne säßen.
Modifiziertes SIR-Modell
[Bearbeiten]Im modifizierten SIR-Modell wird lediglich die Gruppe der Susceptibles vollständig erfasst. Die Infected hingegen beziehen hier nur den Teil an Infizierten ein, welche getestet und erfasst sind. Hierfür wird der konstante Faktor eingeführtt, welcher den prozentualen Anteil der getesteten Infizierten beschreibt. Zudem wird eine neue Infektionsrate mit als Infektionsrate aus dem SI-Modell eingeführt. Die Sterberate wird ebenfalls eingeführt und macht aus den Removed (alte R) die Recovered (neue R). Diese umfassen nun nur noch die Genesenen. Die Toten spielen in diesem Modell nur indirekt eine Rolle. Für und folgt daraus zudem das klassische SIR-Modell.
Die Anfangsbedingungen sehen wie folgt aus:
Die Gesamtpopulation wird sich in diesem Modell insgesamt verringern und zwar um die Anzahl der Verstorbenen. Nach der Erhaltungsgleichung gilt: .
Skript
[Bearbeiten]t_0=0;
b=1000; %Bruchteil z.B. Tausendstel
B=55000000/b; % Kapazitätsgrenze, 2/3 der deutschen Bevölkerung
I_0=16/1000; %Anfangsinfizierte, I(t_0)
S_0=B-I_0; % Anfällige
R_0=0; %Genesene + Tote zu Beginn
Im nächsten Schritt werden die Parameter festgelegt:
k=0.324550 % Infektionsrate
w=1/14; % Wechselrate zu den Genesenen
d=0.003 % Todesrate
r=0.2 % Anteil der erfassten Infizierten
%%aktuelle Daten: (passen nicht mehr zu exponenzieller Funktion)
A=coronaData(); %Funktion coronaData wird aufgerufen
I=A(1,:); %Infected
Rem=A(2,:); %Removed
R=A(3,:); %Recovered
I_aktuell=I-R; %aktuelle Infected
t_I=[t_0:1:length(I)-0.1];
Die Daten des RKI werden nun als erstes geplottet:
figure(1)
plot (t_I,I./b,'s',"linewidth",4,t_I,I_aktuell/b,'*',t_I,R/b,'s',t_I,Rem./b,'*')
hold on
title ('Corona-Daten Deutschland, Quelle RKI')
legend ("Infected (kummuliert)","Infected (aktuell)","Recovered","Removed","location", "west")
grid on
xlabel ('Tage ab dem 26. Februar')
ylabel ('Zahlen in Tausend')
xticks([10,20,30,40,50,60,70,80])
axis([0,80, 0 ,220])
hold off

Im nächsten Schritt soll eine Slowdown-Infektionsrate ermkittelt werden, welche sich der tatsächlichen infektionsrate aus den Daten des RKI annähert. In diesem Fall wurde zur Ermittlung das Augenmaß verwendet.
function val=slowdown1(x,t) %angepasst an Werte des RKI nach Augenmaß
% Ab Tag t (Argument der Funktion) wir die Infektionsrate in mehreren Schritten sinken, x ist Zeitpunkt in Abhängigkeit zu Tag t (5 Tage später, 20 Tage später...)
% Auf Grund von Schulschließung, Kontaktverbot, Maskenpflicht, ...
if x<t val=0.85 ;
elseif x<t +5 val=t./(x.^1.1);
elseif x<t +10 val=t./(x.^1.15);
elseif x<t +22 val=t./(x.^1.25);
elseif x<t +40 val=t./(x.^1.45);
elseif x<t +50 val=t./(x.^1.55);
else val=t./(x.^1.60);
endif
endfunction
function val=slowdown2(x,t)
if x<t val=1.0 ;
elseif x<t +5 val=t./(x.^1.1);
elseif x<t +10 val=t./(x.^1.15);
elseif x<t +22 val=t./(x.^1.2);
elseif x<t +40 val=t./(x.^1.3);
elseif x<t +50 val=t./(x.^1.2);
else val=t./(x.^1.1);
endif
endfunction
function val=slowdown3(x,t)
if x<t val=0.89 ;
elseif x<t +5 val=t./(x.^0.9);
elseif x<t +10 val=t./(x.^1.05);
elseif x<t +22 val=t./(x.^1.2);
elseif x<t +40 val=t./(x.^1.3);
elseif x<t +42 val=t./(x.^1.8);
elseif x<t +50 val=t./(x.^1.4);
else val=t./(x.^1.4);
endif
endfunction
%Zeitabhängige Infektionsrate der Slowdown-Funktionen
for i=1:length(t_I)
k1(i)=slowdown1 (t_I (i), 25)*k;
k2(i)=slowdown2 (t_I (i), 25)*k;
k3(i)=slowdown3 (t_I (i), 25)*k;
endfor
%Infektionsrate aus den RKI-Daten berechnet:
T=1
kk1(1)=k1(1);
for i=T+1:length(I)
kk1(i-T)=(I(i)-I(i-T))/(I(i-T));
endfor
figure (2)
plot (t_I,k1,'m',t_I,k2,'r',t_I,k3,'b')
hold on
plot (kk1,'k*')
grid on
title ('Tatsächtliche und Slowdown-Infektionsraten')
legend ('Slowdown1-Funktion','Slowdown2-Funktion','Slowdown3-Funktion', 'Infektionsrate RKI-Daten' )
hold off
%Lösung des ODE Systems
t=(0:0.1:180);
%Anfangsbedingungen
yo=[S_0;I_0;R_0];
f=@(y,x) [-k*slowdown1(x,25)*y(1)*y(2)/(r*B);k*slowdown1(x,25)*y(1)*y(2)/B-w*y(2);w*y(2)-0*y(2)];
g=@(y,x) [-k*slowdown2(x,25)*y(1)*y(2)/(r*B);k*slowdown2(x,25)*y(1)*y(2)/B-w*y(2);w*y(2)-0*y(2)];
h=@(y,x) [-k*slowdown3(x,25)*y(1)*y(2)/(r*B);k*slowdown3(x,25)*y(1)*y(2)/B-w*y(2);w*y(2)-0*y(2)];
y1= lsode (f, yo, t);
y2= lsode (g, yo, t);
y3= lsode (h, yo, t);
%relat. Fehler Infizierte
%Interpolation der Funktionswerte an die RKI-Zeiten (Tage)
P_f=interp1 (t, y1(:,2), t_I+1);
P_g=interp1 (t, y2(:,2), t_I+1);
P_h=interp1 (t, y3(:,2), t_I+1);
rel_Pf=abs(P_f-I_aktuell./1000)./(I_aktuell./1000);
rel_Pg=abs(P_g-I_aktuell./1000)./(I_aktuell./1000);
rel_Ph=abs(P_h-I_aktuell./1000)./(I_aktuell./1000);
%relat. Fehler Genesene/Toten
Q_f=interp1 (t,y1(:,3),t_I+1);
Q_g=interp1 (t,y2(:,3),t_I+1);
Q_h=interp1 (t,y3(:,3),t_I+1);
rel_Qf=abs(Q_f-R./1000)./(R./1000);
rel_Qg=abs(Q_g-R./1000)./(R./1000);
rel_Qh=abs(Q_h-R./1000)./(R./1000);
figure (3)
plot (t_I,rel_Pf,'m',t_I,rel_Pg,'r',t_I,rel_Ph,'b')
title ('Relativer Fehler I')
legend('f','g','h')
figure (4)
title ('Relativer Fehler R')
hold on
plot (t_I,rel_Qf,'m',t_I,rel_Qg,'r',t_I,rel_Qh,'b')
legend('f','g','h')
axis([35 80 0 1])
figure (5)
title('Vergleich Slowdown1 und RKI-Daten')
hold on
plot (t,y1(:,2),'r.',t,y1(:,3),'c.',t,y1(:,3)+y1(:,2),'b.')
hold on
plot (t_I,I./b,'bs',"linewidth",4,t_I,I_aktuell./b,'r*',t_I,R./b,'c*')
legend ("Infected","Removed","Infected+Removed","German data I+R","German data I","German data R","location","west")
axis([0,80, 0 ,220])
figure (6)
title('Vergleich Slowdown3 und RKI-Daten')
hold on
plot (t,y2(:,2),'r.',t,y2(:,3),'c.',t,y2(:,3)+y2(:,2),'b.')
hold on
plot (t_I,I./b,'bs',"linewidth",4,t_I,I_aktuell./b,'r*',t_I,R./b,'c*')
legend ("Infected","Removed","Infected+Removed","German data I+R","German data I","German data R","location","west")
axis([0,80, 0 ,220])
figure (7)
title('Vergleich Slowdown2 und RKI-Daten')
hold on
plot (t,y3(:,2),'r.',t,y3(:,3),'c.',t,y3(:,3)+y3(:,2),'b.')
hold on
plot (t_I,I./b,'bs',"linewidth",4,t_I,I_aktuell./b,'r*',t_I,R./b,'c*')
legend ("Infected","Removed","Infected+Removed","German data I+R","German data I","German data R","location","west")
axis([0,80, 0 ,220])
Im linken Bild ist deutlich zu erkennen, dass die Slowdown3-Funktion die beste Annährung an die Daten des RKI hat. Wird jedoch der relative Fehler (Mitte) der Infected betrachtet, so fällt auf, dass in etwa bis Tag 5 die Slowdown1-Funktion den geringsten Fehler aufweist und bis etwa Tag 27 die Slowdown2-Funktion den niedrigsten relativen Fehler hat. Ab Tag 30 jedoch gibt es insgesamt einen viel größeren Unterschied in den Fehlermaßen der drei Funktionen. Hier ist die Slowdown3-Funktion ganz eindeutig die beste Wahl. Dies spiegelt sich auch deutlich in den Recovered wieder. Hier liegt der relative Fehler größtenteils sogar im Intervall . Es ist zu beachten, dass die Zeit hier erst ab Tag 30 betrachtet wird, da es zuvor noch keine Genesenen gibt.
Nun wird noch untersucht, welche Auswirkungen Lockerungen nach sich ziehen würden. Hierzu wird eine neue Funktion definiert:
function val=lockerung(x,t)
if x<t val=0.89 ;
elseif x<t +5 val=t./(x.^0.9);
elseif x<t +10 val=t./(x.^1.05);
elseif x<t +22 val=t./(x.^1.2);
elseif x<t +30 val=t./(x.^1.3);
elseif x<t +35 val=t./(x.^1.5);
elseif x<t +45 val=t./(x.^1.2);
elseif x<t +55 val=t./(x.^1.1);
else val=t./(x.^1.0);
endif
endfunction
Es folgt noch die Untersuchung der Testrate :
r1=0.1;
r2=0.3;
r3=0.75;
r4=1.0;
%Anfangswerte
yo=[B-I_0;I_0;R_0;Rem_0];
r11=@(y,x) [-k*y(1)*y(2)/(r1*B);k*y(1)*y(2)/B-w*y(2);w*y(2)-d*y(2);d*y(2)];
r12=@(y,x) [-k*y(1)*y(2)/(r2*B);k*y(1)*y(2)/B-w*y(2);w*y(2)-d*y(2);d*y(2)];
r13=@(y,x) [-k*y(1)*y(2)/(r3*B);k*y(1)*y(2)/B-w*y(2);w*y(2)-d*y(2);d*y(2)];
r14=@(y,x) [-k*y(1)*y(2)/(r4*B);k*y(1)*y(2)/B-w*y(2);w*y(2)-d*y(2);d*y(2)];
y1= lsode (r11, yo, t);
y2= lsode (r12, yo, t);
y3= lsode (r13, yo, t);
y4= lsode (r14, yo, t);
Wie der Animation deutlich zu entnehmen ist, führt ein erhöhter Wert von dazu, dass die Zahl der erfassten Infizierten steigt und die bekannten Fälle näher bis exakt (für ) an die tatsächliche Anzahl an COVID-19 Erkrankungen heranreicht. Für eine kleine Testrate bedeutet dies im Umkehrschluss, dass es eine sehr hohe Dunkelziffer gibt. Diese ist bereits zu erahnen, wenn man die Steigung der Susceptibles betrachtet. Der Anstieg an Infected scheint hier bei kleinem im Vergleich nicht zusammenzupassen, da die Anfälligen viel schneller und stärker abfallen, als die Infizierten ansteigen.

Optimierung
[Bearbeiten]In diesem Modell ist ist die Rate der Datenerfassung konstant. Tatsächlich ist es jedoch der Fall, dass die Testkapazität im Laufe der Pandemie deutlich gestiegen ist und die Anzahl wöchentlich variiert. Geburten und Tod in anderen Zusammenhängen werden ebenfalls nicht mit einbezogen.
Finite Differenzenmethode: Dirichlet und Neumann Randwertprobleme
[Bearbeiten]Dirichlet Randwerte
[Bearbeiten]Bei einem Dirichlet Randwertproblem wird die gesuchte Funktion am Rand durch eine Funktion festgelegt.
Um die gesuchte Funktion zu berechnen, muss eine Approximation für gefunden werden. In einer Raumdimension kann hier betrachtet werden. Die zweite Ableitung kann dabei mit Hilfe des zentralen Differenzenquotienten diskretisiert werden.
- für jedes
(wobei )
Im zweidimensionalen Raum müssen sowhl x als auch y betrachtet werden. Aus diesem Grund werden zwei Differenzenquotienten und aufgestellt.
Diese werden durch die folgenden partiellen Ableitungen im Punkt approximiert:
- ,
Damit ergibt sich:
Um nun eine Systemmatrix aufzustellen zu können, müssen zunächst die unbekannten Funktionswerte aller Gitterpunkte, sowie die Funktionswerte der rechten Seite in je einen langen Vektor eingeordnet werden. Diese sollen folgendermaßen (in der selben Reihenfolge) aufgestellt werden:
Damit kann nun ein lineares Gleichungssystem aufgestellt werden, wobei unsere blockdiagonale Matrix ist. Dabei gilt:
- mit Diagonalblock
und der Einheitsmatrix auf der Nebendiagonale.
Skript
[Bearbeiten]Zuerst muss ein Gitter mit äquidistanten Punkten erstellt werden.
%Endpunkte
xend=8;
yend=8;
%Schrittweite
N=xend*yend;
hx=xend/(N+1);
hy=hx;
M=floor(yend/hy)-1;
%Gitter erstellen
[x,y]=meshgrid(hx:hx:xend-hx,hy:hy:yend-hy);
Anschließend wird der Diffusionskoeffizient festgelegt, sowie die Randbedingungen. Die Dirichlet-Randbedingungen sagen aus, dass die gesuchte Funktion am Rand durch eine gegebene Funktion festgelegt. In diesem Fall werden konstante Randwerte benutzt.
a=1.0; %konstanter Diffusionskoeffizient (beschreibt die Verbreitungsgeschwindigkeit; je größer, desto schneller)
%Dirichlet RB:
unten=0.05;
oben=-0.05;
links=0.01;
rechts=0.01;
V=ones(N*M,1); % erzeugt Vektor der Länge N*M
BB=diag(-4.*V,0)+diag(ones(N*M-1,1),1)+ diag(ones(N*M-1,1),-1)+diag(ones(N*(M-1),1),N)+diag(ones(N*(M-1),1),-N);
%Systemmatrix N^2xM^2 mit -4 auf der Diagonale und 1 auf den Nebendiagonalen und 1 auf N und -N-ter Nebendiagonale
%Korrektur der Matrix (Blockdiagonalität)
for i=1:(M-1)
BB(i*N+1,i*N)=0;BB(i*N,i*N+1)=0;
endfor
%Matrix mit Diffkoeffizient a/h^2 multiplizieren
BB=BB*a/hx^2;
%Matrixdarstellung
figure (1);
spy (BB);
xlabel("Spalten")
ylabel("Zeilen")
zlabel("Matrix B")
title ("Systematrix B");
%Speichern der Bilder (optional):
test=["Systematrix B.jpg"]
saveas(1, test)
function wert=Q(x,y)
if sqrt((x.-5).^2+(y.-6).^2)<=0.25 wert=1;
else wert=0;
endif
endfunction
for i=1:N
for j=1:M
f(j,i)=1*Q(x(j,i),y(j,i));
%Dirichlet Randbedingung unten und oben:
if j==1 f(j,i)=1*Q(x(j,i),y(j,i))+unten*a/hx^2;
elseif j==M f(j,i)=1*Q(x(j,i),y(j,i))+oben*a/hx^2;
%Dirichlet Randbedingung links und rechts:
elseif i==1 f(j,i)=1*Q(x(j,i),y(j,i))+links*a/hx^2;
elseif i==N f(j,i)=1*Q(x(j,i),y(j,i))+rechts*a/hx^2;
endif
endfor
endfor
f(1,1)=1*Q(x(j,i),y(j,i))+(links+unten)*a/hx^2;
f(1,N)=1*Q(x(j,i),y(j,i))+(rechts+unten)*a/hx^2;
f(M,1)=1*Q(x(j,i),y(j,i))+(links+oben)*a/hx^2;
f(M,N)=1*Q(x(j,i),y(j,i))+(rechts+oben)*a/hx^2;
b=-1*reshape(f',N*M,1); %Muss transporniert werden, damit die Zeilen untereinander aufgestellt werden
%Lösungsschritt
sol=BB\b;
sol_matrix=reshape(sol,N,M);% Matrix mit N(Zeilen)x M(Spalten)
sol_matrix=sol_matrix'; % Rücktransponierung nach reshape
figure(2);
surfc(x,y,sol_matrix);
colormap winter;
title ("Lösung");
ylabel("y")
xlabel("x")
%Speichern der Bilder (optional):
test=["Lösung.jpg"]
saveas(2, test)
figure(3);
surfc(x,y,f);
title ("Quellfunktion Q");
ylabel("y")
xlabel("x")
%Speichern der Bilder (optional):
test=["Quellfunktion Q.jpg"]
saveas(3, test)
Plot
[Bearbeiten]
Werte unten=0.05; oben=-0.05; links=0.01; rechts=0.01;
%Dirichlet RB: unten=-0.05; oben=0.05; links=0.05; rechts=-0.01;
Gemischte Randwerte
[Bearbeiten]Bei der Verknüpfung vob Dirichlet- und Neumann-Randbedingungen müssen die Richtungsableitungen der Funktion in Richtung des äußeren Normalenvektors gegeben sein.
Im zweidimensionalen Raum ergibt sich oder . In dieser Aufgabe wird die Finite-Differenzen-Methode (FDM) für Neumann-RB auf dem Rechteck D mit homogenen Randbedingungen - - implementiert. Allgemein gilt jedoch:
- linker Rand:
- rechter Rand:
- unterer Rand:
- oberer Rand:
Skript
[Bearbeiten]%Endpunkte
xend=10;
yend=10;
%Schrittweite
N=xend*yend;
hx=xend/(N+1);
hy=hx;
M=floor(yend/hy)-1;
N=N+2;
M=M+2;
%Gitter erstellen
[x,y]=meshgrid(0:hx:xend,0:hy:yend);
a=1.0 %konstanter Diffusionskoeffizient (beschreibt die Verbreitungsgeschwindigkeit; je größer, desto schneller)
%Dirichlet RB:
unten=0.05;
oben=-0.05;
V=ones(N*M,1); % erzeugt Vektor der Länge N*M
BB=diag(-4.*V,0)+diag(ones(N*M-1,1),1)+ diag(ones(N*M-1,1),-1)+diag(ones(N*(M-1),1),N)+diag(ones(N*(M-1),1),-N);
%Systemmatrix N^2xM^2 mit -4 auf der Diagonale und 1 auf den Nebendiagonalen und 1 auf N und -N-ter Nebendiagonale
%Korrektur der Matrix (Blockdiagonalität)
for i=1:(M-1)
BB(i*N+1,i*N)=0;BB(i*N,i*N+1)=0;
endfor
%Matrix mit Diffkoeffizient a/h^2 multiplizieren
BB=BB*a/hx^2;
h=hx;
for i=1:(M-2)
v_up=zeros(1,N*M);
v_up(N*i+1)=a/h;
v_up(N*i+2)=-a/h;
v_down=zeros(1,N*M);
v_down(N*i+N)=a/h;
v_down(N*i+N-1)=-a/h;
BB(i*N+1,:)=v_up ;
BB(i*N+N,:)=v_down ;
endfor
#auf linkem/rechten Rand Neumann Randbedingungen
function wert=Q(x,y)
if sqrt((x.-4).^2+(y.-3).^2)<=0.55 wert=1;
else wert=0;
endif
endfunction
for i=1:N
for j=1:M
f(j,i)=1*Q(x(j,i),y(j,i));
%Neumann Randbedingung links und rechts:
if i==1 f(j,i)=1*Q(x(j,i),y(j,i)); endif
if i==N f(j,i)=1*Q(x(j,i),y(j,i)); endif
%Dirichlet Randbedingung unten und Oben:
if j==1 f(j,i)=1*Q(x(j,i),y(j,i))+unten*a/hx^2; endif
if j==M f(j,i)=1*Q(x(j,i),y(j,i))+oben*a/hx^2; endif
endfor
endfor
%Ecken haben Mittelwert aus anliegenden Rändern
f(1,1)=1*Q(x(j,i),y(j,i))+0.5*unten*a/hx^2;
f(1,N)=f(1,1);
f(M,1)=1*Q(x(j,i),y(j,i))+0.5*oben*a/hx^2;
f(M,N)=f(M,1);
b=-1*reshape(f',N*M,1); %Muss transporniert werden, damit die Zeilen untereinander aufgestellt werden
%Lösungsschritt
sol=BB\b;
sol_matrix=reshape(sol,N,M);% Matrix mit N(Zeilen)x M(Spalten)
sol_matrix=sol_matrix'; % Rücktransponierung nach reshape
Plot
[Bearbeiten]figure (1);
spy (BB);
xlabel("Spalten")
ylabel("Zeilen")
zlabel("Matrix B")
title ("Systematrix B");
%Speichern der Bilder (optional):
test=["Systematrix B.jpg"]
saveas(1, test)
figure(2);
surfc(x,y,sol_matrix);
colormap winter;
title ("Lösung");
ylabel("y")
xlabel("x")
%Speichern der Bilder (optional):
test=["Lösung1.jpg"]
saveas(2, test)
figure(3);
surfc(x,y,f);
title ("Quellfunktion Q");
ylabel("y")
xlabel("x")
%Speichern der Bilder (optional):
test=["Quellfunktion Q1.jpg"]
saveas(3, test)
Aufgabe 5: Anwendung am Beispiel Landau
[Bearbeiten]Anmerkung
[Bearbeiten]Diese Aufgabe entstand in Zusammenarbeit mit Gruppe 1.
Beispiel Landau
[Bearbeiten]In der letzten Tutoriumsaufgabe wird das Finite-Differenzen-Verfahren zur Lösung der Reaktionsdiffusionsgleichung für die zeitlich-räumliche Modellierung der Corona-Ausbreitung auf einem rechteckigen Gebiet D implementiert. In unserem Fall modellieren wir die Corona-Ausbreitung für die Stadt Landau. Die Informationen bezüglich der Fläche und Bevölkerungsdichte von Landau erhalten wir unter folgendem Link: Landau in der Pfalz Wir betrachten in unserer Modellierung die Bevölkerungsdichte für die Kernstadt Landau.
Fläche: 83 km2 (ungefähre Quadratsfläche: 9 km *9 km) Bevölkerungsdichte in Landau Zentrum: 2619,9 Einwohner pro km2
Von Runkelbold - Eigenes Werk, CC BY-SA 3.0, https://commons.wikimedia.org/w/index.php?curid=3663743
Festlegung des Gebietes als Gitter
[Bearbeiten]In der Abbildung oben kann man erkennen, dass Landau geographisch grob als Quadrat gesehen werden kann. Mit der bekannten Fläche können dann damit die Kantenlängen festgelegt werden.
xend=9.0; %in km
yend=9.0; %in km
N=60; % Anzahl von Gitterpunkten in x-Richtung(0-te und N-te Index entsprechen den Rändern, keine Gitterpunkte am Rand)
a=0.01; %konstanter Diffusionskoeffizient %Beschreibt die Verbreitungsgeschwindigkeit(Je größer, desto schneller)
T=100; %Zeitintervall in Tagen
t=0.03; %Zeitschritt
Um das Gitter zu konstruieren, muss die Kantenlänge der Quadrates durch geteilt werden, um die Gitterpunkte in gleichgroßem Abstand auf der Kante zu verteilen, bzw. den Abstand der Gitterpunkte zu berechnen. Da ein Quadrat vorliegt, ist die Anzahl der Gitterpunkte in x- und y-Richtung gleich groß.
hx=xend/(N+1) %Abstand Gitterpunkte
hy=hx; % setze so, damit sich Quadrate bilden
%M=floor((yend/hy))-1 -> Anzahl der Gitterpunkte in y-Richtung
M=N; %Im Falle eines Quadrates
times=floor (T/t); %Anzahl der Zeitschritt
Parameter für die Reaktionsdiffusionsgleichung
[Bearbeiten]B=2619,9; %Bevölkerungsdichte Einwohner pro Quadratkilometer(Landau Zentrum)
k=0.254; %Infektionsrate aus SIR-Abgabe3 (siehe Residuum Gruppe 3)
w=1/14; % Wechselrate
Definition der Anfangsfunktion
[Bearbeiten]function wert=initialfunction(x,y)
wert=0;
r=0.25; %Fläche des Kreises: 0.20km^2entspricht ca. 523 Einwohnern
if sqrt((x.-4.5).^2+(y.-4.5).^2)<=r wert=1/(pi*r^2); %Infektionen im Zentrum von Landau (ein Infizierter)
endif
endfunction
Systemmatrix
[Bearbeiten]Das erstellte Gitter muss nun um die Randpunkte erweitert werden.
[x,y]=meshgrid(0:hx:xend,0:hy:yend);
N=N+2;
M=M+2;
Die folgende Systemmatrix wird nach dem gleichen Schema wie in Aufgabe 4 erstellt.
V=ones(N*M,1); % erzeugt vektor der Laenge N*M
BB=diag(-4.*V,0)+diag(ones(N*M-1,1),1)+ diag(ones(N*M-1,1),-1)+diag(ones(N*(M-1),1),N)+diag(ones(N*(M-1),1),-N);
% Systemmatrix N^2xN^2 mit -4 auf der Diagonale und 1 auf den Nebendiagonalen und 1 auf den N und -N-ter Nebendiagonale
for i=1:(M-1)
BB(i*N+1,i*N)=0;BB(i*N,i*N+1)=0; %Korrektur der Blockmatrix (Blockdiagonalität)
endfor
BB=BB*a/hx^2;
Festlegen der Neumann Randbedingungen
Im folgenden sollen die Neumann-Randbedingungen festgelegt werden. Dabei sollen die jeweiligen Ableitungen der Ränder null sein. Dies beschreibt einen freien Fluss der Infizierung in die angerenzenden Gebiete. Für Neumann Randbedingungen auf dem oberen und unteren Rand müssen in unserer Systemmatrix die erste und letzte Blockzeile angepasst werden.
% Neumann RB für y: a* partial_y u=0 - einzelne Blöcke der Matrix ersetzen
h=hx;
block=diag((a/h)*ones(N,1)); % Matrix mit h bzw. -h auf Diagonale
%Unterer Rand
BB(1:N,1:N)=-block;
BB(1:N,N+1:2*N)=block;
%Oberer Rand
BB(N*(M-1)+1:N*M,N*(M-1)+1:N*M)=-block;
BB(N*(M-1)+1:N*M,N*(M-2)+1:N*(M-1))=block;
Für Neumann Randbedingungen auf dem linken und rechten Rand müssen die oberste und unterste Zeile unserer Diagonalblöcke von BB ersetzt werden.
% Neumann RB für x: a* partial_x u=0 - einzelne Zeilen der Matrix ersetzen
for i=1:(M-2)
v_up=zeros(1,N*M);
v_up(N*i+1)=a/h;
v_up(N*i+2)=-a/h;
v_down=zeros(1,N*M);
v_down(N*i+N)=a/h;
v_down(N*i+N-1)=-a/h;
BB(i*N+1,:)=-v_up; %Vektor der kompletten ersten Zeile
BB(i*N+N,:)=-v_down ; %Vektor der kompletten letzten Zeile
endfor
Graphische Darstellung der Systemmatrix
figure (67);
spy (BB);
xlabel("Spalten")
ylabel("Zeilen")
zlabel("Matrix B")
title ("Systematrix B");

Anfangslösung als Matrix
[Bearbeiten]Durch den Parameter I0 ist die Wahl der Anfangsinfizierungen variabel. Wir gehen in diesem Fall von einem Infizierten im Zentrum von Landau aus.
I0=1 %Anzahl der Anfangsinfizierungen pro gegebene Fläche pi*r^2
for i=1:N
for j=1:M
initial(j,i)=I0*initialfunction(x(j,i),y(j,i));
endfor
endfor
size (initial)
sol_old=1*reshape(initial',N*M,1);
%reshape funktioniert Spaltenmäßig: Sp1,Sp2, wir wollen Zeilenmäßig Matrix f' ist N(Zeilen) x M (Spalten)
Einlesen der Graphik zur Einwohnerzahl
[Bearbeiten]In diesem Abschnitt wird eine Graphik zur Einwohnerzahl als Bevölkerungsmatrix eingelesen. Danach wählen wir einen für Landau geeigneten Bildausschnitt.
A=imread ('Monitor_Einwohnerzahl_detail.png');
size(A); % Zeigt die Dimension der Bildmatrix
imagesc(A(5:13,4:12)) % Bildanzeige
S=A(5:13,4:12);
S=double (S);
[s1 s2]=size(S);
Max=max(max(S))
S=Max-S; %Farbumkehrung/Anpassung
Interpolation der Werte an die Koordinatenmatrix
[Bearbeiten]Die oben aufgestellte Matrix S ist eine 9 x 9 Matrix. Diese Matrix ist allerdings gröber eingeteilt, als unser vorher definiertes Gitter mit viel kleineren Schritten. Das bedeutet, dass wir die Werte von S auf unsere Gitterpunkte interpolieren müssen. Dabei verwenden wir eine zweidimensionale kubische Interpolation.
Sint=interp2(S,xend*(1*x.+2)/(xend+2),yend*(1*y.+2)/(yend+2), 'cubic');
Sint=flipud (Sint); %Bild flippen oben <-> unten
size (Sint)
figure(66);
surface(x,y, (B/Max)*Sint)
title ("Bevoelkerungsdichte");
ylabel("y")
xlabel("x")
colorbar
B=reshape((B/Max)*Sint',N*M,1).+0.1*Max; %Angepasste Bevölkerungsdichte
size (B)
Bild links: Von Runkelbold - Eigenes Werk, CC BY-SA 3.0, https://commons.wikimedia.org/w/index.php?curid=3663743
Zwischenfazit Die Gegenüberstellung der beiden Bilder zeigt, dass wir einen geeigneten Ausschnitt unserer Bildmatrix für die Bevölkerungsdichte in Landau gewählt haben. Die Bevölkerungsdichte ist im Zentrum (Kernstadt Landau) am größten und wird in den umliegenden Ortschaften deutlich geringer.
Teil 1: Lösungsschritt - Reaktionsterm für kumulierte Infizierte
[Bearbeiten]Im ersten Teil der Implementierung stellen wir einen Reaktionsterm für die kumulierten Infizierten auf, das heißt, dass wir Infizierte und Genesene betrachten (einmal infiziert, immer infiziert)
Reaktionsterm für kumulierte Infizierte
Im nachfolgenden Reaktionsterm wird die Bevölkerungsdichte geographisch differenziert als Funktion einer Raumvariable betrachtet:
F=@(u) (k./B).*u.*(B.-u);
Dieser Term wird nun zu unserem System von gewöhnlichen Differentialgleichungen des räumlich diskretisierten Neumann-Randwertproblems für die Diffusion hinzugefügt.
Würde man die homogene Differentialgleichung betrachten gilt folgendes:
Dabei lässt sich hier das Stern-Approximationsschema anwenden:
Aus diesem ursprünglichen System ergibt sich also , welches nun zur Reaktionsdiffusionsgleichung ergänzt werden kann:
- .
Zeitschleife
Der numerische Lösungsschritt (Zeitdiskretisierung) erfolgt hier mithilfe des Expliziten Eulerverfahrens: Nach der Anwendung des Vorwärtsdifferenzenquotienten erhält man folgende Berechnungsformel: , die sich in der implementierten Zeitschleife widerspiegelt:
for i=1:times
sol= sol_old+ BB*sol_old*t + 1*F(sol_old)*t;
sol_old=sol;
matrix_solution(:,i)=sol;
endfor
Auswahl an Bildern - jedes fünfte Bild
fig_index=floor([0.05:0.05:1]*times)
j=0
for i=fig_index
sol_matrix=reshape(matrix_solution(:,i),N,M);%Matrix mit N(Zeilen) x M(Spalten)
sol_matrix=sol_matrix';
disp(['Figure ',num2str(round(i/fig_index(1)))]);
j=j+1;
figure(j);
surface(x,y,sol_matrix*h^2, "EdgeColor", "none") %multipliziert mit hx^2, damit Anzahl der Infizierten pro Raster und nicht Dichte dargestellt wird
colormap ("jet")
axis([0 xend 0 yend 0 1.1*max(max(B))*hx^2])
colorbar
title (["Loesung in t=", num2str(round(delta_t*i))]);
ylabel("y")
xlabel("x")
test=["Fig_", num2str(j),".jpg"]
endfor
Anfangslösung
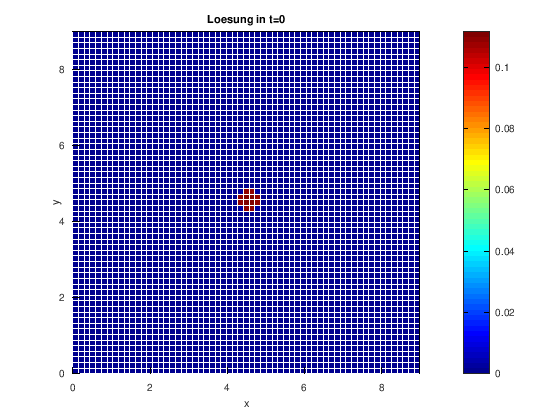
Die Abbildung zeigt, dass wir zu Beginn der Corona-Ausbreitung ca. einen Infizierten im Zentrum haben.
Animation

Teil 2: Lösungsschritt - Reaktionsterm für aktuell Infizierte
[Bearbeiten]In diesem Teil möchten wir die gleiche Implementation wie oben durchführen, aber nun anstatt der kumulierten Infizierten die aktuell Infizierten betrachten. Hierfür benötigen wir unsere Kenntnisse aus dem SIR-Modell. Dabei wird unser SIR-Modell auf die räumliche Verbreitung ausgeweitet, indem der Diffusionsterm hinzugefügt wird. Unsere Funktionen heißen deshalb nicht mehr nur S, I und R, sondern u_S bzw. u_I (u_R wird an dieser Stelle ignoriert) und sind nicht mehr nur von der Zeit t abhängig, sondern auch von einer Raumvariable.
Es werden zwei gekoppelte Reaktionsdiffusionsgleichungen für die Dichtefunktionen gelöst. Die entsprechenden Reaktionsterme sehen wie folgt aus:
Vorbereitung der alten Lösung
sol_old=1*reshape(initial',N*M,1); %Aktuell Infizierte
sol_old_susp=B.-sol_old; %Infektionsanfällige
Definition der Slowdown-Funktion
Wir benötigen für den Reaktionsterm eine Slowdown-Funktion. Diese übernehmen wir aus unserer Implementierung des SIR-Modells aus der dritten Tutoriumsaufgabe.
function val=slowdown3(x,t)
if x<t val=1 ;
elseif x<t +8 val=t./(x.^1.1);
elseif x<t +16 val=t./(x.^1.25);
elseif x<t +22 val=t./(x.^1.4);
else val=t./(x.^1.5);
endif
if x> 61 val=0.5 ; endif %Lockerungen
endfunction
Reaktionsterm aktuell Infizierte
Nun definieren wir eine nichtlineare Funktion von u (aktuell Infizierte), v (Empfängliche) und der Zeit t.
F=@(u,v,t) (k*slowdown3(25,t)./B).*u.*v;
Zeitschleife
Der Zeitdiskretisierung erfolgt an dieser Stelle auch wieder mithilfe des expliziten Eulerverfahrens.
for i=1:times
Reakt=F(sol_old,sol_old_susp, i*delta_t); %Nichtlinearer Term, wird aus alten Werten berechnet in der Form F=@(u,v,t)
sol_susp= sol_old_susp+ BB*sol_old_susp*delta_t - Reakt*delta_t; %Erster Reaktionsterm: Empfängliche
sol= sol_old+ BB*sol_old*delta_t+ (Reakt-w.*sol_old)*delta_t; %Zweiter Reaktionsterm: Aktuell Infizierte (um Term -w*I erweitert)
sol_old=sol; %Speicherung für den nächsten Zeitschritt (alte Lösung wird auf neue Lösung überschrieben)
sol_old_susp=sol_susp;
matrix_solution1(:,i)=sol; %Speicherung der aktuell Infizierten in einer Matrix, Speicherung der Anfälligen (S) nicht implementiert
endfor
Animation
Anschließend erhalten wir mit dem gleichen Octave-Befehl wie im ersten Teil unsere Animation für die aktuell Infizierten.
fig_index=floor([0.05:0.05:1]*times)
j=0
for i=fig_index
sol_matrix=reshape(matrix_solution1(:,i),N,M);% Matrix mit N(Zeilen)x M(Spalten)
sol_matrix=sol_matrix';
disp(['Figure ',num2str(round(i/fig_index(1)))]);
j=j+21;
figure(j);
% Alternativ surfc(x,y,sol_matrix*h^2, "EdgeColor", "none") -> 3D
surface(x,y,sol_matrix*h^2, "EdgeColor", "none") % multipliziert mit hx^2, damit Anzahl der Infizierten pro Rasterdargestellt wird (und nicht die Dichte)
colormap ("jet")
axis([0 xend 0 yend 0 1.1*max(max(B))*h^2])
colorbar
title (["Aktuelle Infizierte Lösung in t=", num2str(round(t*i))]);
ylabel("y")
xlabel("x")
%test=["Aktuelle Infizierte Fig_", num2str(j),".jpg"]
%saveas(j, test)
endfor
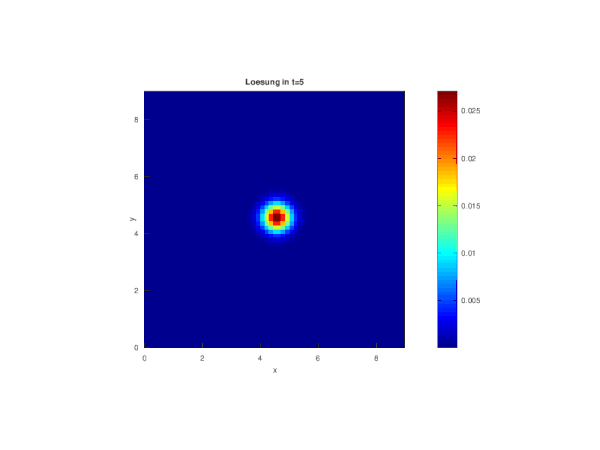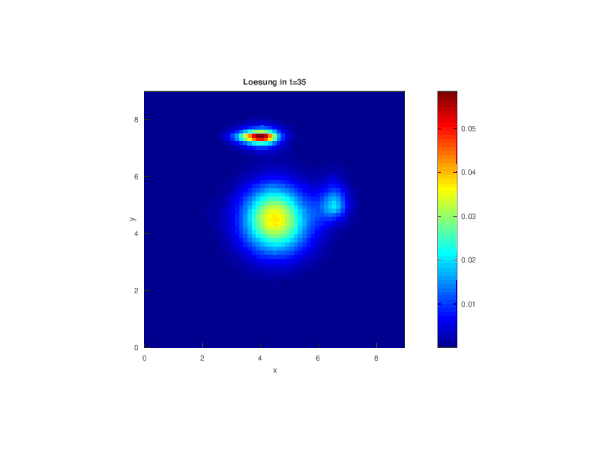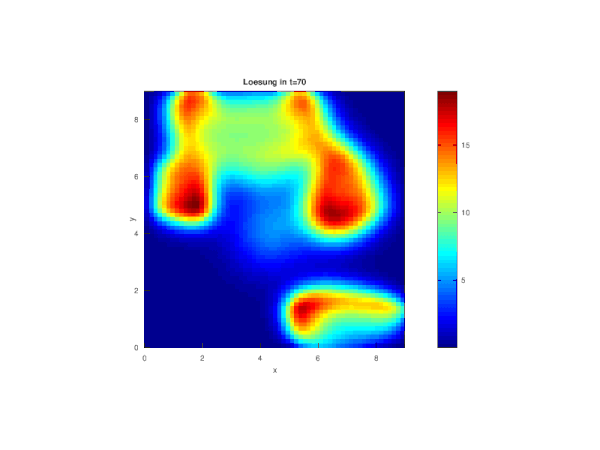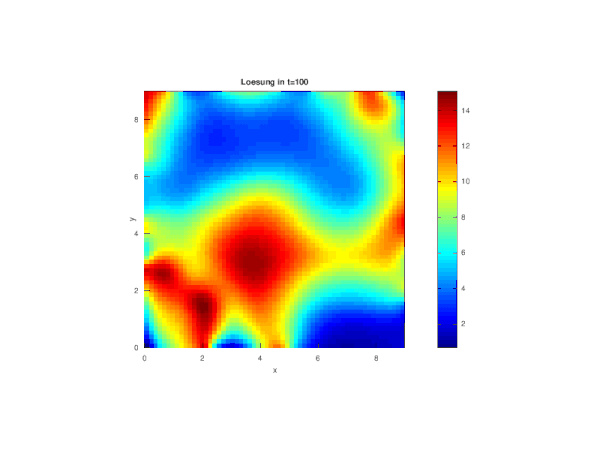
-->
Funktionsdefinitionen
[Bearbeiten]Bitte passen die Beschreibung für Ihre definierten Funktionen an:
Transportprozess zwischen Zellen im Raum
[Bearbeiten]Name der Funktion : Die Funktion übernimmt die Aufgabe ...
Mathematische Beschreibung: Transportprozess
[Bearbeiten]- Definitionsbereich der Funktion:
- Wertebereich der Funktion:
- Definition der Funktion in mathematischer Notation:
- oder ggf. mit Fallunterscheidung
Implementation der Funktion: Transportprozess
[Bearbeiten]Die Funktion wurde mit der Syntax von Octave implementiert: Name der Funktion :
- Definition der Funktion
function Sneu = transProc(pS) % die Funktion beschreibt den epidemiologischen Prozess in einer Zelle Sneu = ... endfunction
- Aufruf der Funktion
S = [2,1;3,2;30,2]
S =
2 1
3 2
30 2
transProc(S)
Epidemiologischer Prozess in jeder Zelle
[Bearbeiten]Name der Funktion : Die Funktion übernimmt die Aufgabe ...
Mathematische Beschreibung: Epidemiologischer Prozess
[Bearbeiten]- Definitionsbereich der Funktion:
- Wertebereich der Funktion:
- Definition der Funktion in mathematischer Notation:
- oder ggf. mit Fallunterscheidung
Implementation der Funktion: Epidemiologische Prozess
[Bearbeiten]Die Funktion wurde mit der Syntax von Octave implementiert:
function Sneu = epiProc(pS) % die Funktion beschreibt den epidemiologischen Prozess in einer Zelle Sneu = ... endfunction
- Aufruf der Funktion
S = [2,1;3,2;30,2]
S =
2 1
3 2
30 2
epiProc(S)
Ergänzungen Octave-Tutorial
[Bearbeiten]Diese Ergänzungen wurden bei dem Octave-Tutorial vorgenommen, um die Implementation der Gruppe bzgl. der verwendeten Octave-Befehle nachvollziehbar zu machen.
- Matrix-Multiplikation in Octave - notwendig für die Modellierung diskreter Transportprozesse.
Skripte in Octave
[Bearbeiten]Referenzieren Sie hier die von Ihnen verwendeten Skripte und Bibliotheken in Octave oder R/Studio
Literatur
[Bearbeiten]Notieren Sie hier die von Ihnen verwendete Literatur
- Boto von Querenburg (2013) Mengentheoretische Topologie - Springer-Lehrbuch










![{\displaystyle [1,N_{t}]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/0808875c3e0716d07a045809c1ff331d08cb3720)









![{\displaystyle [0,50]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/b44dc494d31dd5e8cf7784ae3beb7730d7da8bce)







![{\displaystyle n\in [80,99]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/c24b139052feb0ac5ff39270ddc2ea655b1c47f9)




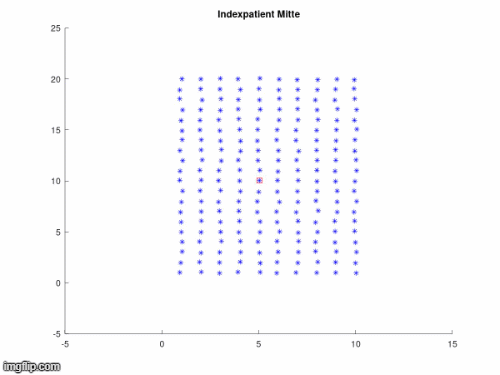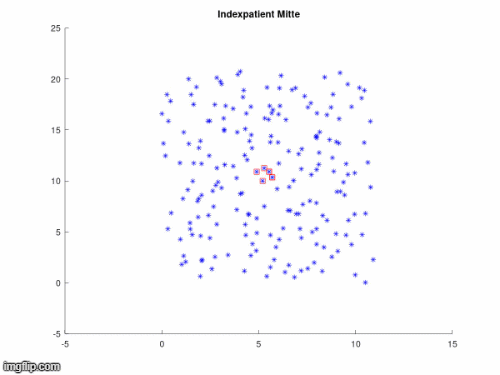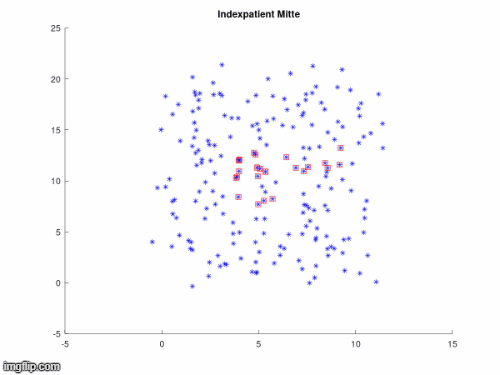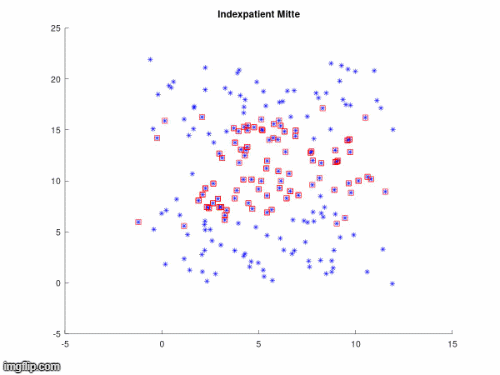

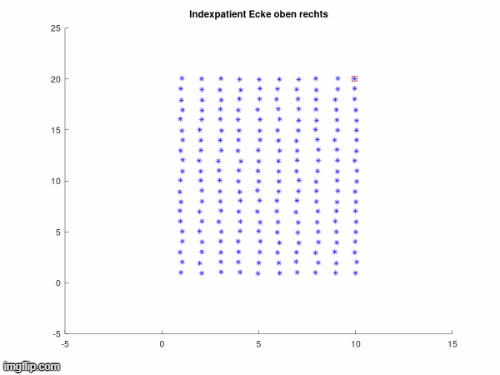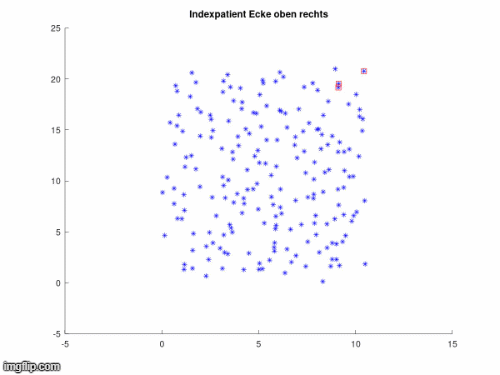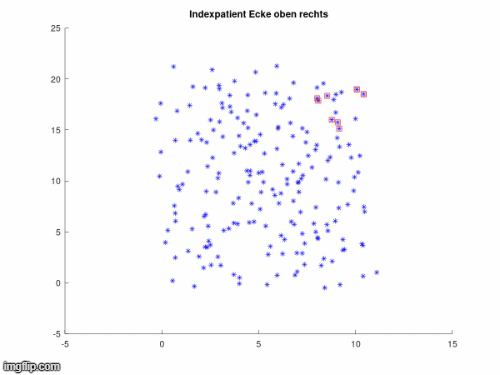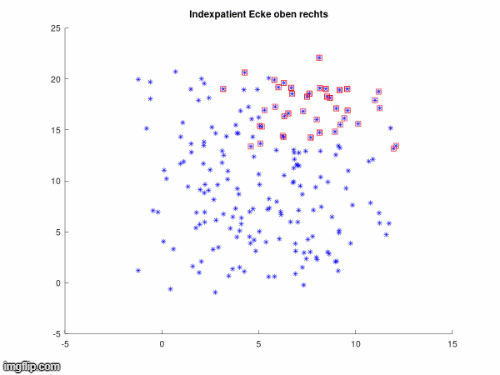


















































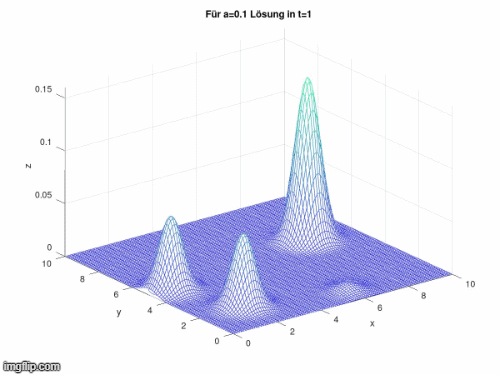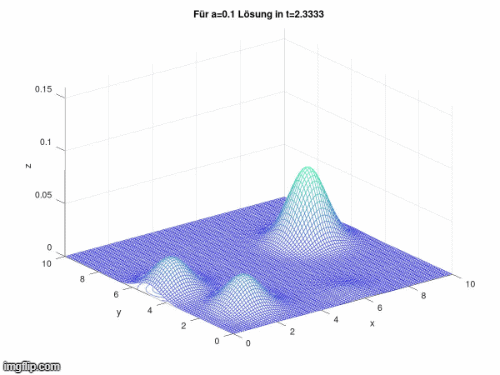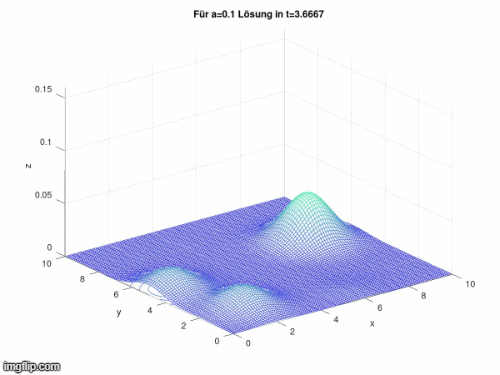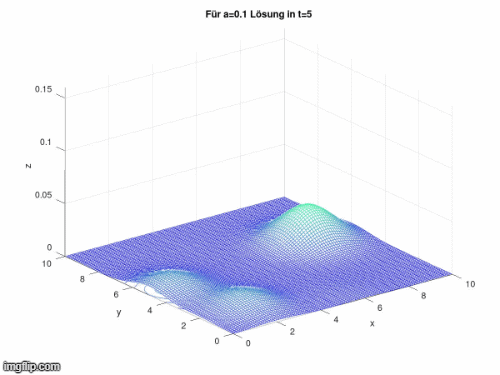



















































![{\displaystyle [0,0.1]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/8b836c8608550b3f9099d9139d54844c3ff2ac90)








![{\displaystyle r\in [0,1]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/6d4a8acea9f5c4e59d8f5fd0ea3c695efa7252fe)





























































![{\displaystyle {\begin{array}{rcl}transProc:\mathbb {D} &\to &\mathbb {W} \\(S,x)&\mapsto &\left\{{\begin{array}{lcl}1&{\mbox{ für }}&x_{1}\in [a,b]\\0&{\mbox{ für }}&x_{1}\notin [a,b]\\-1&{\mbox{ sonst }}&\end{array}}\right.\\\end{array}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/e0a1f4951bfd24fa7ba5939f3e759aaaa3c08811)

